Introducción
la mortalidad de lechones es uno de los principales rasgos de selección en la cría de cerdos y está influenciada por la cerda, los lechones y el medio ambiente. Por lo tanto, la mortalidad de los lechones es un fenotipo complejo y depende de la capacidad de la cerda para criar a su descendencia, pero también es una función del peso al nacer, el manejo y la selección (Knol et al., 2002)., Sin embargo, también los defectos recesivos monogénicos contribuyen a la mortalidad de los lechones, aunque solo se han reportado pocos ejemplos en el pasado (Murgiano et al., 2012; Matika et al., 2019). Incluso en aquellos casos en los que el efecto de la mutación es grave, la selección eficiente contra tal mutación se ve obstaculizada por la baja frecuencia. En muchos defectos graves, los cigotos mueren muy temprano en la gestación, dejando ningún rastro más que la ausencia de homocigotos en la población en general(Derks et al., 2019).,
Los efectos de la endogamia en las poblaciones de cerdos comerciales generalmente se mantienen bajo control mediante la cría selectiva para la disminución de la mortalidad en lechones al mejorar tanto las capacidades maternas como la viabilidad de los lechones (Olijslagers, 2018). Sin embargo, las variantes subyacentes a los defectos monogénicos recesivos no se capturan bien dentro de los valores genéticos, y potencialmente se desplazan a frecuencias más altas como resultado de una selección intensa (Georges et al., 2019). Además, esas variantes también pueden mantenerse como resultado de la selección de equilibrio para un efecto positivo correlacionado en estado heterocigoto (Derks et al., 2018).,
Los defectos recesivos solo contribuyen marginalmente a la mortalidad global de los lechones (Alonso-Spilsbury et al., 2007). Sin embargo, las variantes que afectan a la mortalidad de los lechones son de gran importancia porque esas variantes influyen directamente en la producción y el bienestar animal (Baxter et al., 2013; Rutherford et al., 2013). Sin embargo, en el manejo de la población animal, la ocurrencia de defectos de baja frecuencia generalmente está mal documentada (a menudo se usan términos muy generales), y los síndromes a menudo solo se reconocen una vez que han alcanzado una frecuencia alta., Esto es especialmente relevante para los síndromes que no conducen a fenotipos muy distintos. Por lo tanto, incluso en las poblaciones de reproducción comercial, se puede hacer poco seguimiento de síndromes específicos, y para seleccionar eficazmente contra síndromes específicos de baja frecuencia, por lo tanto, requiere nuevos enfoques.
en este trabajo, describimos el descubrimiento de un síndrome altamente debilitante en una población porcina comercial a través de una encuesta basada en una combinación de matrices SNP de densidad media y secuenciación del genoma completo (WGS)., La encuesta condujo a la identificación de una deleción de desplazamiento de marco de 16 PB en el gen SPTBN4, con claras consecuencias fenotípicas predichas en homocigotos. La frecuencia de portadoras es de alrededor del 9% en la población en estudio, afectando aproximadamente al 0,81% de las camadas de la población. La frecuencia era lo suficientemente baja como para ser desconocido para tener una base genética, e incluso efectivamente no ser reconocido como un síndrome específico en absoluto. Tras la realización de la encuesta, se identificó una cerda preñada engendrada por un jabalí portador., Los lechones afectados sufren de miopatía y son incapaces de caminar, lo que generalmente resulta en la muerte a las pocas horas después del nacimiento, completamente en línea con la patología predicha en comparación con casos similares en humanos y ratones.
Material y métodos
Animales, genotipos y preprocesamiento
el conjunto de datos consta de 31.839 animales de una línea sintética de jabalí con fondo blanco grande. La línea es mantenida y criada en las granjas Topigs Norsvin nucleus, principalmente seleccionando características de producción y salud., Los animales fueron genotipados en el chip Illumina GeneSeek custom 50K SNP (Lincoln, NE, USA). Se eliminaron animales con una frecuencia de genotipos faltantes > 0,15. Se descartaron los marcadores que no cumplían los siguientes criterios de filtrado: una tasa de llamada mínima de 0,85, una frecuencia de alelo menor > 0,01, y una prueba exacta de proporciones de Hardy-Weinberg p-valor por debajo de p < 10-12. Además, se descartaron marcadores con ubicación desconocida en la estructura del genoma de Sscrofa11.1, dejando 41.573 marcadores después del filtrado., Todos los pasos se realizaron en Plink v1. 90b3.30 (Purcell et al., 2007).
Haplotype Phasing and Identification of SSC6 haplotype
se realizó el haplotype phasing and imputation of missing sites in Beagle5.0 con el parámetro para el tamaño efectivo de la población establecido en 100, otros ajustes fueron predeterminados (Browning et al., 2018). Los homocigotos esperados se estimaron basándose en la frecuencia de los haplotipos, utilizando el principio de Hardy-Weinberg. Se aplicó una prueba binomial exacta para probar el número de homocigotos observados con el número de homocigotos esperados., El haplotipo se consideró significativamente agotado si P < 5 × 10-3.
efectos fenotípicos asociados con el haplotipo SSC6
se examinaron los registros del haplotipo SSC6 sobre el número total de nacidos, el número de nacidos muertos, los lechones momificados, la supervivencia en el parto y la supervivencia en la lactancia (supervivencia hasta aproximadamente 21 días de edad) de un total de 9.666 camadas. Enumeramos estos fenotipos para todas las camadas CXC y CXN identificadas. Se utilizó una prueba T de Welch para evaluar si los fenotipos de las camadas CxC difieren significativamente de las camadas CxN. Un valor p < 0.,05 fue considerado significativo.
análisis de secuenciación del genoma completo e identificación de variantes candidatas
el conjunto de datos consta de 71 individuos secuenciados del genoma completo de la población en estudio. Las 71 muestras también estuvieron presentes en nuestro conjunto de datos de 31,839 animales genotipados en el 50K. las 71 muestras tienen un volumen total de 1.93 Tbp (pares de bases de tera) de 14.16 mil millones de lecturas de extremos pares de 150-bp (tabla S3). Las muestras fueron secuenciadas en Illumina HiSeq 2000. Alineamos las secuencias a la construcción del genoma de Sscrofa11.1 usando la versión 0.7 de BWA-MEM.,15 (Li y Durbin, 2009) con una mapabilidad media del 98,9% y una cobertura muestral que oscila entre 8,8 y 14,8 X (10,9 x promedio). Samblaster se utilizó para eliminar duplicados de PCR (Faust y Hall, 2014). Samtools se utilizó para ordenar, combinar e indexar archivos bam (Li et al., 2009). Se generaron mapas y estadísticas de calidad utilizando Qualimap (Okonechnikov et al., 2016). La llamada de variantes se realizó con Freebayes v1.1. 0 con los siguientes ajustes: –min-base-quality 10 –min-alternate-fraction 0.2 –haplotype-length 0 –min-alternate-count 2 (Garrison y Marth, 2012)., Se descartaron variantes con puntuación de calidad Phred < 20 (Li et al., 2009). Las variantes fueron anotadas usando el predictor de efecto de variante Ensembl (VEP, release 96) (Mclaren et al., 2016). El impacto de las variantes sin sentido se predijo utilizando la clasificación de intolerantes de tolerantes (Sift) (Kumar et al., 2009). El análisis de DL se realizó utilizando Plink v1.90b3. 30 (Purcell et al., 2007) con los siguientes ajustes –chr-set 18, –r2, LD-window-r2 0.8.
alineación de la proteína SPTBN4
La alineación de la proteína entre el tipo salvaje y la proteína mutante se realizó usando ClustalO (Madeira et al.,, 2019) y visualizado usando Espcript 3 (Robert y Gouet, 2014). Se realizó una visualización y validación adicional utilizando el visor del genoma JBrowse versión 1.12.1 (Skinner et al., 2009).
la validación de la deleción SPTBN4 Causal de 16 PB
la PCR se realizó utilizando 60 ng de ADN genómico, con 0.4 µm de cada imprimación, 1.8 mM MgCl2 y 25 unidades / ml de Onetaq® DNA Polymerase (OneTaq® 2x Master Mix with Standard Buffer, New England Biolabs) en el buffer PCR del fabricante en un volumen final de 12 µl., La desnaturalización inicial durante 1 min a 95 ° C fue seguida de 35 ciclos de 95°C para 30 s, 55°C para 45 s, 72°C 90 s, seguido de una extensión de 5 min 72°C. los cebadores de PCR para SPTBN4 son TCAAGGGTGCAGGCTCTTC forward y ggtaggaagctcgaagtggg reverse. El primer delantero fue teñido-etiquetado con cualquiera de 6-FAM para producir un producto de PCR fluorescente etiquetado detectable en Abi 3730 secuenciador de ADN (Applied Biosystems). Los tamaños de los fragmentos se determinaron utilizando el software GeneMapper 5 de ABI.,
examen histopatológico
dos lechones afectados de menos de 1 semana de edad fueron enviados al Departamento de patología de Royal Animal Health (Deventer) para su examen. Macroscópicamente, todas las observaciones estuvieron dentro de los límites normales. El músculo esquelético de la pata delantera, el músculo dorsal y la pata trasera de ambos animales se muestreó para la tinción rutinaria H&E y PTAH. El tejido muscular se almacenó en frascos separados y se fijó en solución de formaldehído al 4%, tamponada (=solución de formalina al 10%, tamponada)., Después de eso, el tejido se incrustó en parafina y se cortó en 2 µm de acuerdo con el procedimiento de operación estándar (SOP RAH). Posteriormente, los portaobjetos fueron desparafinados y teñidos rutinariamente para detectar hematoxilina y eosina (h&E) en una máquina automática de color. Simultáneamente se prepararon portaobjetos adicionales de 2 µm del tejido muscular, así como un portaobjeto de control positivo del tejido muscular para la tinción manual con «hematoxilina de ácido fosfotungstico», abreviado como PTAH. Esta tinción se prefiere para demostrar estrías cruzadas del músculo esquelético.,
valores genéticos y análisis de Asociación
en este estudio, se evaluaron 63 rasgos utilizados en el programa de mejoramiento. Los valores genéticos estimados desregresados (DEBV) se utilizaron como variable de respuesta para cada rasgo en estudio. El valor genético estimado (VEB) de todos los rasgos evaluados fue desregresado utilizando la metodología descrita por Garrick et al. (2009). El VEB de cada animal se obtuvo a partir de la evaluación genética rutinaria realizada por un programa de cría comercial (Topigs Norsvin) utilizando un modelo animal., Las fiabilidades por animal para el propósito de la desregulación se extrajeron de la evaluación genética basada en la metodología de Tier y Meyer (2004). Las heritabilidades utilizadas para la desregulación también se extrajeron de la evaluación genética rutinaria. Finalmente, los factores de ponderación basados en la fiabilidad estimada de la DEBV también se estimaron de acuerdo con Garrick et al. (2009) usando un valor de 0.5 para el escalar C. para asegurar la calidad del DEBV, solo animales con un factor de ponderación mayor que cero y una confiabilidad del DEBV mayor que 0. ,20 fueron utilizados en los análisis de asociación. La confiabilidad de la DEBV también se obtuvo de acuerdo con Garrick et al. (2009).
los análisis de asociación se realizaron utilizando el software ASREML (Gilmour et al.,, 2009) aplicando el siguiente modelo animal mixto lineal:
donde DEBVij es el DEBV observado para el animal j, w es el factor de ponderación para el residual, µ es la media general de DEBV de la población, Ri es el estado portador (recuento del alelo perjudicial) de la mutación 4 i, aj es el efecto genético aditivo estimado utilizando una matriz de relación media basada en pedigrí y el error residual. Las asociaciones con a-log10 (valor de P) mayor que cinco fueron declaradas como significativas.
resultados
A 1.,El segmento de 5 Mb en el cromosoma 6 afecta la supervivencia de la lactancia en cerdos
analizamos 31.638 animales de una sola línea de jabalí de raza pura (línea sintética con fondo blanco grande), genotipada en el chip SNP porcino 50K (construcción Sscrofa11.1) (Warr et al., 2019). El análisis reveló un segmento de 1,5 Mb en el cromosoma 6 (SSC6:48,75–50,25) que muestra un déficit de homocigosidad asociado con una reducción de la supervivencia de la lactancia (Tablas 1 y 2). El haplotipo se está segregando a una frecuencia alélica moderada del 4,5% (9,0% de frecuencia portadora) en la población en estudio., La frecuencia de los haplotipos ha fluctuado en la última década, pero disminuyó en los últimos 3 años (figura S1). Probamos si la frecuencia fue impulsada por un efecto de ventaja heterocigota. Sin embargo, encontramos asociaciones mayoritariamente negativas con rasgos de selección importantes, a excepción de la profundidad del lomo y la longitud de gestación (Tabla 3), lo que sugiere que la frecuencia es puramente el resultado de la deriva genética.

la Tabla 1 SSC6 haplotipo características.,

Tabla 2 las camadas portadoras por camadas muestran una disminución del 24% en la supervivencia de la lactancia en comparación con las camadas portadoras por camadas no portadoras. Los resultados significativos se indican en negrita.

la Tabla 3 Rasgos asociados significativamente con portadores heterocigotos de la SPTBN4 eliminación.
Las 52 camadas portador por portador (CxC) no muestran una reducción significativa en el número total de animales nacidos o nacidos vivos., Sin embargo, la supervivencia de la lactancia se reduce en aproximadamente un 24% en camadas CxC en comparación con los apareamientos portadores por no portadores (CxN), lo que indica que los lechones homocigotos mueren dentro del período de lactancia (Tabla 2). A continuación, examinamos los comentarios sobre el tiempo y la causa de mortalidad de las camadas de CxC. Esto reveló que la mayoría de los lechones que murieron dentro de las primeras 24 h después del nacimiento. La mayoría de esos lechones fueron descritos por los agricultores como » lechones débiles al nacer.,»
El análisis de secuenciación del genoma completo revela una deleción de 16 bp Frameshift en SPTBN4 como la variante causal probable
para identificar la mutación causal, examinamos los datos de la secuencia del genoma completo de 71 animales de la población bajo estudio e identificamos cinco animales portadores. El análisis de desequilibrio de enlace (DL) reveló 267 Variantes de SNP e indel en DL alta (r2 > 0.8) con el haplotipo SSC6 (tabla S1), la mayoría en DL perfecta (247 variantes)., Solo cinco variantes potencialmente afectan la secuencia de codificación (tres missense, un frameshift, un Splice-acceptor). Se predice que las tres variantes missense serán toleradas por SIFT (score > 0.18, tabla S1), mientras que la variante splice-acceptor afecta a un gen que codifica un péptido de 28 bp de función desconocida, poco probable que sea causal. Sin embargo, se predijo que una variante en DL completa (r2 = 1) con el haplotipo tendría un alto impacto; una deleción de desplazamiento de marco de 16 PB en el exón 26 del gen SPTBN4 (6:g.48801280delGACGGTGTACGCCGGT) (Figuras 1A, B)., La deleción frameshift (ENSSSCP00000031537:P. Arg1902fs) introduce 30 nuevos aminoácidos y un codón de parada prematura, produciendo una proteína 4 no eritrocítica beta (SPTBN4) disminuida y truncada. Los mutantes carecen de los 662 aminoácidos finales de la proteína de tipo salvaje (figura 1C), incluido el dominio de homología de pleckstrin (PH) requerido para el transporte de proteínas a las membranas (Wang et al., 2018). La proteína SPTBN4 es un miembro de las proteínas beta-espectrinas y es una actina que une la membrana celular con el citoesqueleto de actina., Las mutaciones de SPTBN4 interrumpen la maquinaria citoesquelética que controla la localización adecuada de los canales iónicos en los nervios mielinizados causando neuropatías motoras (Parkinson et al., 2001; Wang et al., 2018).

la Figura 1 (A) SPTBN4 modelo gen. La ubicación del exón 26 afectado está indicada en rojo. (B) ilustración de la supresión de 16-bp. La figura muestra el tipo salvaje y el exón mutante. C) alineación de la secuencia de proteínas mutantes (Mt) y de tipo salvaje (Wt) SPTBN4. La mutación induce 30 nuevos aminoácidos y un codón de parada prematuro.,
el genotipado de cinco camadas de CxC confirma la deleción de SPTBN4 como el probable culpable
genotipamos cinco camadas de CxC para la deleción de 16-bp que tuvo al menos dos lechones (rango 2-6) que murieron dentro de las primeras 48 h después del nacimiento. Las cinco camadas produjeron 53 lechones de los cuales 19 eran homocigotos para la deleción de 16 PB (Tabla 4). Los 19 lechones homocigotos murieron dentro de las 48 h después del nacimiento (18 dentro de las 24 h). De los 34 lechones restantes (8 de tipo salvaje y 26 portadores), solo 1 murió dentro de las 48 h, probablemente causada por otros factores (ambientales).,

Tabla 4 Genotipado de la eliminación de frameshift SPTBN4 causal probable de 16 PB en cinco camadas portador por portador. La suma por clase de genotipo se indica en negrita.
los lechones homocigotos para la deleción de SPTBN4 sufren de miopatía y parálisis de las extremidades posteriores
monitoreamos una camada reciente de CxC (fecha de parto: 28 de abril de 2019) que produjo seis lechones sanos, dos afectados (muestras: 9912, 9916) (figura 2a) y tres lechones nacidos muertos., Se confirmó el estado de deleción homocigótica de SPTBN4 para los dos lechones afectados (tabla S2). Además, se observaron cuatro portadores heterocigotos y dos lechones homocigotos de tipo salvaje entre los individuos sanos. Uno de los lechones nacidos muertos (muestra: 9921) también era homocigoto para la deleción, mientras que los otros dos eran heterocigotos. Los lechones afectados sufren de debilidad muscular extrema (Figuras 2B, C), parálisis de las extremidades posteriores y temblores (Video S1). Por lo tanto, los lechones no podían caminar ni beber.,
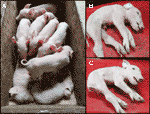
la Figura 2 (A) Dos lechones afectados (en vivo) junto con seis saludable compañeros de camada. Los lechones derivan de un apareamiento CXC parido el 28 de abril de 2019. B) lechón macho afectado 9912. C) lechón hembra afectada 9916.
los lechones afectados carecen de estriaciones cruzadas en los músculos esqueléticos dorsales y de las extremidades posteriores
el examen histopatológico reveló degeneración dispersa de las fibras musculares en ambos lechones, y necrosis y vasculitis focales en el músculo dorsal en uno de los lechones (ID = 9912)., Además, la tinción con ácido fosfotungstico hematoxilina (PTAH) muestra coloración divergente de las fibras del músculo esquelético, lo que indica una disminución de las estrías cruzadas, particularmente en los músculos de las patas dorsales y traseras de los animales afectados (figura 3b), mientras que las patas delanteras parecen no estar afectadas (figura 3a). La disminución de las estrías cruzadas está indicada por coloración anormal y pérdida general de volumen de fibras musculares (figura 3B). Los cambios histopatológicamente observados en las patas traseras y en los músculos dorsales son indicativos de distrofia muscular.,

Figura 3 (a) vista transversal de un músculo esquelético desde la pierna delantera. La flecha negra indica la coloración normal (oscura) de las fibras musculares, lo que indica la presencia de estrías cruzadas. PTAH Bar = 50 µm. B) vista transversal de un músculo esquelético desde la pata trasera. La flecha negra indica una coloración anormal (rosa) de las fibras musculares, lo que indica la falta de estrías cruzadas. La flecha amarilla indica coloración normal y presencia de estrías cruzadas. PTAH Bar = 50 µm.,
discusión
en este trabajo reportamos un nuevo defecto congénito causante de mortalidad de lechones probablemente debido a una deleción de desplazamiento de marco de 16 PB en el gen SPTBN4. Los lechones sufren de debilidad muscular extrema (miopatía) y mueren pocas horas después del nacimiento. Se espera que la deleción confiera una pérdida completa de la función de la espectrina beta, proteína no eritrocítica 4. SPTBN4 es un miembro de la familia de genes espectrinos y es necesario para la agrupación de canales iónicos en los nodos de Ranvier, afectando el potencial de acción (Devaux, 2010)., Las mutaciones interrumpen la maquinaria citoesquelética que controla la localización adecuada de los canales iónicos y la función de los dominios axonales principalmente en los segmentos iniciales del axón (AIS) y los nodos de Ranvier (Wang et al., 2018). Más específicamente, el dominio C-terminal afectado de SPTBN4 es crucial para el tráfico de canales KCNQ2 y la excitabilidad en los nodos de Ranvier (Devaux, 2010).
La investigación de seguimiento posterior identificó casos en humanos y ratones que indicaron que el síndrome resultante probablemente no resultaría inmediatamente letal, sino que conferiría miopatía grave., Mediante datos de genotipo SNP de densidad media, disponibles para todos los animales de la población reproductora (N = 31.839), se pudieron identificar portadores. Entre esas portadoras había una cerda que estaba aproximadamente a medio término en el embarazo en el momento de la identificación, engendrada por un jabalí que también era portadora. Se notificó a la granja de cría que documentara la camada al nacer. El fenotipo observado de los lechones afectados (miopatía, parálisis de las extremidades posteriores, temblores) fue completamente congruente con lo observado en pacientes humanos con pérdida de función homocigótica o mutaciones heterocigóticas compuestas en el gen SPTBN4 (OMIM: 606214)., Dos de los pacientes humanos tienen mutaciones de pérdida de función dentro del dominio de PH (Wang et al., 2018), apoyando que una pérdida del dominio del PH en cerdos probablemente conduciría a una pérdida completa de función de la proteína SPTBN4. En humanos, mutaciones similares conducen a miopatía congénita severa causada por la ausencia de fibras musculares tipo I, neuropatía y sordera (Knierim et al., 2017; Wang et al., 2018). Wang et al. (2018) también observaron neuropatía axonal motora en varios pacientes caracterizada por hipotonía congénita, debilidad profunda y pérdida de reflejos tendinosos profundos en la primera infancia., Además, las biopsias nerviosas revelaron canales nodales na + reducidos y no canales nodales KCNQ2 K+, revelando la patología molecular causante de disfunción del sistema nervioso. Por lo tanto, concluimos que esta variante frameshift es la mutación causal probable que conduce al fenotipo observado y al agotamiento del genotipo homocigoto en la población. Futuros estudios podrían centrarse en hacer un knockout in vivo del gen SPTBN4 en cerdo, para estudiar el síndrome y el fenotipo asociado con más detalle.,
no observamos degeneración de las fibras musculares en las patas delanteras, mientras que las fibras musculares dorsales y traseras se vieron claramente afectadas. Esta observación podría explicar en parte la parálisis de las extremidades posteriores, mientras que las patas delanteras no se ven afectadas. La discrepancia entre las fibras musculares de las patas delanteras y traseras también se ha descrito en ratones temblorosos, en los que las mutaciones de pérdida de función de SPTBN4 causan neuropatía motora, parálisis de las extremidades traseras, temblores y sordera central (Parkinson et al., 2001; Komada y Soriano, 2002). Parkinson et al., (2001) describen velocidades reducidas de conducción nerviosa en nervios ciáticos de ratones con alelos temblorosos que causan la neuropatía periférica de las extremidades posteriores. La expresión de SPTBN4 en ratones está restringida al cerebro, la médula espinal y los nervios ciáticos y no se observa en el músculo esquelético, por lo que esta enfermedad es principalmente un defecto neuronal. En general, no está claro qué mecanismo causa la ausencia de síntomas en las extremidades anteriores. Este «knockout natural» en cerdos puede ser un recurso útil para estudiar la enfermedad humana, ya que los cerdos suelen ser un mejor modelo para estudiar la enfermedad humana en comparación con las especies de roedores., Además, la consecuencia de la pérdida de la función SPTBN4 se puede estudiar con más detalle.
el tamaño efectivo de la población (Ne) de la raza en estudio se estima en alrededor de 100 (Hidalgo et al., 2016). En la cría de animales, La Ne baja aumenta el riesgo de que los alelos perjudiciales aumenten en frecuencia por casualidad. Además, estudios previos han demostrado que los alelos letales recesivos pueden ser impulsados por efectos ventajosos en heterocigotos (Derks et al., 2018; Matika et al., 2019). Matika et al.,, 2019 encontró una mutación recesiva de stop-gained en el gen MSTN asociada con un aumento importante en la profundidad muscular en heterocigotos. Sin embargo, no encontramos evidencia de ninguna ventaja heterocigótica en nuestro estudio. Con las técnicas genómicas actuales, ahora podemos identificar alelos deletéreos que se desplazan a frecuencias más altas, y monitorear la aparición de nuevos alelos deletéreos con precisión, lo que permite una purga más efectiva. Además, el resultado de este tipo de estudio mejorará en gran medida la conciencia de los defectos genéticos «ocultos» tanto a nivel de criador como de agricultor., Sin ninguna información previa, los defectos de nacimiento raros a menudo se registran como » lechón débil.»Y sin ninguna distinción adicional de síndromes específicos, no es posible tomar más medidas. En la mayoría de los casos se desconoce si existe una base genética o si puede haber otros efectos de confusión. Con información genómica previa, el síndrome se puede identificar, en comparación con otros casos, y los portadores identificados, lo que lleva a información procesable.
la mortalidad de los lechones es de gran importancia económica y para el bienestar animal., Por lo tanto, el descubrimiento de la mutación SPTBN4 ha llevado a la implementación inmediata en el programa de reproducción para minimizar la frecuencia de apareamientos portador por portador. Esto permite evitar el nacimiento de los individuos afectados, mejorando así el bienestar animal y reduciendo las pérdidas económicas.
conclusión
en este estudio se reporta un nuevo defecto congénito probablemente causado por una deleción recesiva del cambio de Marco en el gen SPTBN4 en cerdos. Los hallazgos están respaldados por sorprendentes similitudes con los fenotipos sindrómicos asociados a SPTBN4 en humanos y ratones., El estudio permite monitorear y purgar el alelo deletéreo de la población. Se pueden prevenir los cruces de transporte por transporte, excluyendo a los individuos afectados, reduciendo así las pérdidas económicas y mejorando el bienestar animal. Finalmente, estos «knockouts naturales» obtenidos en la industria de la cría pueden proporcionar un modelo para la enfermedad humana y aumentar nuestra comprensión de la función génica dentro del clado de mamíferos, y proporcionar un modelo potencial para la enfermedad humana.,
Declaración de disponibilidad de datos
Declaración ética
no se requirió revisión ética y aprobación para el estudio en animales porque los datos utilizados en este estudio se obtuvieron como parte de la recolección de datos rutinaria de los programas de cría de Topigs Norsvin, y no específicamente para el propósito de este proyecto. Por lo tanto, la aprobación de un comité de ética no era obligatoria. La recogida de muestras y el registro de datos se llevaron a cabo estrictamente de acuerdo con la ley holandesa sobre protección y bienestar animal (Gezondheids – en welzijnswet voor dieren)., Se obtuvo el consentimiento informado por escrito de los propietarios para la participación de sus animales en este estudio.
contribuciones de los autores
MG, H-JM y MD concibieron y diseñaron el estudio. BH fue responsable de la organización general y la comunicación con Topigs Norsvin y los agricultores. MD Y ML realizaron el análisis de los datos. BD y KL realizaron trabajos de laboratorio. SG-V realizó el análisis patológico. MD escribió el manuscrito. H-JM, MG, BH, SG-V, BD, KL y ML proporcionaron comentarios y sugerencias útiles y ayudaron a redactar el manuscrito. Los datos fenotípicos se analizaron por ML., Todos los autores leyeron y aprobaron el manuscrito final.
financiación
Esta investigación fue financiada por la Asociación STW-Breed4Food, proyecto número 14283: de la secuencia al fenotipo: detección de variación perjudicial mediante predicción de funcionalidad. Este estudio fue financiado por NWO-TTW y los Socios de Breed4Food Cobb Europe, CRV, Hendrix Genetics y Topigs Norsvin. Además, este estudio fue apoyado por el proyecto IMAGE (Horizon 2020, No. 677353). Los financiadores no tuvieron ningún papel en el diseño del estudio, la recolección y análisis de datos, la decisión de publicar o la preparación del manuscrito., El uso del cluster HPC fue posible gracias a CATAgroFood (Shared Research Facilities Wageningen UR).
Descargo de responsabilidad
los datos utilizados en este estudio se han obtenido como parte de la recolección de datos rutinaria de los programas de cría de Topigs Norsvin, y no específicamente para el propósito de este proyecto. Por lo tanto, la aprobación de un comité de ética no era obligatoria. La recogida de muestras y el registro de datos se llevaron a cabo estrictamente de acuerdo con la ley holandesa sobre protección y bienestar animal (Gezondheids – en welzijnswet voor dieren).,
conflicto de intereses
ML y BH son empleados del Centro de investigación Topigs Norsvin, un instituto de investigación estrechamente relacionado con uno de los financiadores (Topigs Norsvin).
Todos los demás autores declaran que la investigación se realizó en ausencia de relaciones comerciales o financieras que pudieran ser interpretadas como un potencial conflicto de intereses.
Los otros Socios de Breed4Food Cobb Europe, CRV, Hendrix Genetics, declaran no tener intereses en competencia para este estudio.,
agradecimientos
Los autores agradecen a Frank van Haaren y Toon Janssen por organizar la comunicación con la granja. Agradecemos a Manon Houben su contribución a los exámenes patológicos. Agradecemos a Mout Viller por tomar videos y fotos de los lechones afectados y sanos después del nacimiento. Agradecemos a Jürgen Harlizius por cuidar de los lechones después de la entrega. Agradecemos a Gerda van Eldik la recogida de muestras. Agradecemos a Egbert Knol y Egiel Hanenberg por el apoyo general y la orientación de Topigs-Norsvin.,
material suplementario
Video S1 / Video que muestra a ambos individuos afectados después del nacimiento.
tabla S1 / variación genómica en DL alta con el haplotipo SSC6.
tabla S2 / Genotipado de la camada CxC incluyendo dos individuos afectados (fecha de parto: 28 de abril de 2019). La tabla muestra el estado homocigoto del/del de los dos lechones afectados 9912,9916.
Table S3 / Mapping and coverage statistics from WGS samples.
figura S1 / SPTBN4 frecuencia portadora de eliminación de 2007-2019.