introductie
Biggensterfte is een van de belangrijkste selectiekenmerken in de varkensfokkerij en wordt beïnvloed door de zeug, de biggen en het milieu. Biggensterfte is dus een complex fenotype en hangt af van het vermogen van de zeug om zijn nakomelingen op te voeden, maar is ook een functie van geboortegewicht, management en selectie (Knol et al., 2002)., Echter, ook monogene recessieve defecten dragen bij tot biggensterfte, hoewel er in het verleden slechts enkele voorbeelden zijn gemeld (Murgiano et al., 2012; Matika et al., 2019). Zelfs in die gevallen waar het effect van de mutatie ernstig is, wordt efficiënt selecteren tegen een dergelijke mutatie belemmerd door de lage frequentie. Bij veel ernstige defecten sterven zygoten heel vroeg in de dracht, waardoor er geen sporen achterblijven behalve de afwezigheid van homozygoten in de bevolking in het algemeen (Derks et al., 2019).,
Inteelteffecten bij commerciële varkenspopulaties worden gewoonlijk onder controle gehouden door selectief fokken op een verminderde sterfte bij biggen door zowel de moedercapaciteit als de levensvatbaarheid van biggen te verbeteren (Olijfslagers, 2018). Varianten die ten grondslag liggen aan recessieve monogene defecten zijn echter niet goed vastgelegd binnen de fokwaarden, en kunnen als gevolg van intense selectie naar hogere frequenties afdrijven (Georges et al., 2019). Bovendien kunnen deze varianten ook worden gehandhaafd als gevolg van balancering selectie voor een gecorreleerd positief effect in heterozygote toestand (Derks et al., 2018).,
recessieve defecten dragen slechts marginaal bij aan de totale biggensterfte (Alonso-Spilsbury et al., 2007). Niettemin zijn varianten die van invloed zijn op de biggensterfte van groot belang omdat deze varianten rechtstreeks van invloed zijn op de productie en het dierenwelzijn (Baxter et al., 2013; Rutherford et al., 2013). Bij het beheer van de dierpopulatie is het optreden van defecten met een lage frequentie echter meestal slecht gedocumenteerd (vaak worden zeer algemene termen gebruikt), en syndromen worden vaak pas herkend als ze een hoge frequentie hebben bereikt., Dit is met name relevant voor syndromen die niet tot zeer verschillende fenotypen leiden. Daarom kan zelfs in commerciële broedpopulaties weinig tracking worden gedaan op specifieke syndromen, en om effectief te selecteren tegen specifieke laagfrequente syndromen vereist daarom nieuwe benaderingen.
in dit werk beschrijven we de ontdekking van een zeer slopend syndroom in een commerciële varkenspopulatie door middel van een onderzoek gebaseerd op een gecombineerde medium-density SNP arrays en whole-genome sequencing (WGS)., Het onderzoek leidde tot de identificatie van een 16-bp frameshift deletie in het sptbn4 gen, met voorspelde duidelijke fenotypische gevolgen in homozygotes. De draagfrequentie bedraagt ongeveer 9% in de onderzochte populatie en treft ongeveer 0,81% van de nesten van de populatie. De frequentie was laag genoeg om Onbekend te zijn om een genetische basis te hebben, en zelfs effectief niet herkend als een specifiek syndroom op alle. Bij de uitvoering van het onderzoek werd één drachtige zeug geïdentificeerd als verwekker van een dragerzwijn., De aangetaste biggen lijden aan myopathie en zijn niet in staat om te lopen, wat meestal resulteert in de dood binnen een paar uur na de geboorte, volledig in lijn met voorspelde pathologie in vergelijking met vergelijkbare menselijke en muis gevallen.
materiaal en methoden
Dieren, Genotypes en voorbewerking
de dataset bestaat uit 31.839 dieren uit een synthetische varkenslijn met een grote witte achtergrond. De lijn wordt onderhouden en gefokt in Topigs Norsvin nucleus boerderijen, voornamelijk selecteren op productie-en gezondheidseigenschappen., De dieren werden gegenotypeerd op de Illumina GeneSeek custom 50K SNP chip (Lincoln, NE, USA). Dieren met een frequentie van ontbrekende genotypen > 0,15 werden verwijderd. We hebben markers weggegooid die niet aan de volgende filtercriteria voldeden: een minimale aanroepsnelheid van 0,85, een kleine allelfrequentie > 0,01, en een Hardy-Weinberg proportions exact test P-waarde onder P < 10-12. Bovendien werden de tellers met onbekende plaats op de sscrofa11. 1 genoombouw verworpen, die 41.573 tellers na het filteren achterlieten., Alle stappen werden uitgevoerd in Plink v1.90b3.30 (Purcell et al., 2007).
Haplotype fasering en identificatie van Ssc6 Haplotype
we voerden haplotype fasering en imputatie van ontbrekende sites uit in Beagle5. 0 met parameter voor effectieve populatiegrootte ingesteld op 100, andere instellingen waren standaard (Browning et al., 2018). De verwachte homozygoten werden geschat op basis van haplotype frequentie, gebruik makend van het Hardy-Weinberg Principe. Een exacte binomiale test werd toegepast om het aantal waargenomen homozygoten te testen met het aantal verwachte homozygoten., Het haplotype werd als significant uitgeput beschouwd als P < 5 × 10-3.
fenotypische effecten geassocieerd met Ssc6 Haplotype
we onderzochten het ssc6 haplotype Voor gegevens over het totale aantal geboren, doodgeboren, gemummificeerde biggen, de overleving van het werpen en de lactatie (overleving tot ongeveer 21 dagen oud) van een totaal van 9.666 nesten. We hebben deze fenotypen voor alle CXC en CXN nesten geïdentificeerd. We gebruikten een Welch ‘ s t-test om te beoordelen of de fenotypen uit de CXC nesten significant verschillen van CXN nesten. A p-waarde < 0.,05 werd als significant beschouwd.
geheel Genoomsequentieanalyse en identificatie van Kandidaatvarianten
de dataset bestaat uit 71 geheel genoomsequenced individuen uit de onderzochte populatie. Alle 71 monsters waren ook aanwezig in onze dataset van 31.839 dieren gegenotypeerd op de 50K. de 71 monsters hebben een totaal volume van 1,93 Tbp (tera base pairs) van 14.16 miljard 150-bp gepaarde-end reads (tabel S3). De monsters werden gesequenced op Illumina HiSeq 2000. We hebben de sequenties afgestemd op de sscrofa11.1 genoom build met behulp van BWA-MEM versie 0.7.,15 (Li en Durbin, 2009) met een gemiddelde mappability van 98,9% en een steekproefdekking variërend van 8,8 tot 14,8 X (10,9 x gemiddeld). Samblaster werd gebruikt om PCR-duplicaten te verwijderen (Faust and Hall, 2014). Samtools werd gebruikt voor het sorteren, samenvoegen en indexeren van bam-bestanden (Li et al., 2009). Mapping en kwaliteitsstatistieken werden gegenereerd met behulp van Qualimap (Okonechnikov et al., 2016). Variant calling werd uitgevoerd met Freebayes v1. 1.0 met de volgende instellingen: –min-base-quality 10 –min-alternate-fraction 0.2 –haplotype-length 0 –min-alternate-count 2 (Garrison and Marth, 2012)., Varianten met Phred quality score < 20 werden verworpen (Li et al., 2009). Varianten werden geannoteerd met behulp van de Ensembl variant effect predictor (VEP, release 96) (McLaren et al., 2016). De impact van missense varianten werd voorspeld met behulp van sorting intolerant from tolerant (SIFT) (Kumar et al., 2009). LD-analyse werd uitgevoerd met Plink v1.90b3.30 (Purcell et al., 2007) met de volgende instellingen –chr-set 18, –r2, ld-window-R2 0.8.
SPTBN4 Eiwituitlijning
eiwituitlijning tussen het wildtype en het mutanteiwit werd uitgevoerd met behulp van ClustalO (Madeira et al.,, 2019) en gevisualiseerd met behulp van ESPript 3 (Robert en Gouet, 2014). Verdere visualisatie en validatie werd uitgevoerd met behulp van de jbrowse genome viewer versie 1.12.1 (Skinner et al., 2009).
validatie van causale 16 bp SPTBN4 deletie
PCR werd uitgevoerd met 60 ng genomisch DNA, met 0,4 µm van elke primer, 1,8 mM MgCl2 en 25 eenheden/ml OneTaq® DNA-Polymerase (OneTaq® 2X Master Mix with Standard Buffer, New England Biolabs) in de PCR-buffer van de fabrikant in een eindvolume van 12 µl., De eerste denaturatie gedurende 1 min bij 95°C werd gevolgd door 35 cycli van 95°C gedurende 30 s, 55 ° C gedurende 45 s, 72 ° C gedurende 90 s, gevolgd door een verlenging van 5 min 72°C. PCR-primers voor SPTBN4 zijn tcaagggtggcaggctcttttc vooruit en ggtaggaagctcgaagtggg achteruit. De voorwaartse inleiding werd dye-geëtiketteerd met of 6-FAM om een fluorescently geëtiketteerd PCR-product te produceren detecteerbaar op ABI 3730 DNA-sequencer (Toegepaste Biosystems). Fragmentgrootte werd bepaald met behulp van GeneMapper software 5 van ABI.,
histopathologisch onderzoek
twee getroffen biggen jonger dan 1 week werden voor onderzoek naar de afdeling pathologie van Koninklijke diergezondheid (Deventer) gestuurd. Macroscopisch waren alle waarnemingen binnen de normale grenzen. De skeletspieren van de voorpoot, de rugspier en de achterpoot van beide dieren werden bemonsterd voor routine h&e kleuring en PTAH kleuring. Het spierweefsel werd opgeslagen in afzonderlijke potten en gefixeerd in formaldehydeoplossing 4%, gebufferd (=formalineoplossing 10%, gebufferd)., Daarna werd het weefsel ingebed in paraffine en in 2 µm gesneden volgens de standaardbehandelingsprocedure (SOP RAH). Daarna werden de objectglaasjes gedeparaffiniseerd en routinematig gekleurd voor hematoxyline en eosine (H&E) in een automatische kleurenmachine. Tegelijkertijd werden extra dia ‘ s van 2 µm van het spierweefsel evenals een positieve controledia van spierweefsel voorbereid voor de handmatige kleuring met “phosphotungstic acid hematoxylin,” afgekort als PTAH. Deze kleuring heeft de voorkeur voor het aantonen van dwarsverbindingen van skeletspieren.,
fokwaarden en Associatieanalyse
in deze studie hebben we 63 eigenschappen geëvalueerd die in het fokprogramma werden gebruikt. Gederegresseerde geschatte fokwaarden (DEBV) werden gebruikt als responsvariabele voor elke eigenschap in studie. De geschatte fokwaarde (EBV) van alle geëvalueerde eigenschappen werden gedereguleerd met behulp van de methodologie beschreven door Garrick et al. (2009). De EBV van elk dier werd verkregen uit de routine genetische evaluatie door een commercieel fokprogramma (Topigs Norsvin) met behulp van een diermodel., De betrouwbaarheid per dier met het oog op de deregulering is ontleend aan de genetische evaluatie op basis van de methodologie van Tier and Meyer (2004). De erfenissen die voor de deregulering werden gebruikt, werden ook uit de routinematige genetische evaluatie gehaald. Tot slot, wegingsfactoren op basis van de geschatte betrouwbaarheid van de DEBV werden ook geschat volgens Garrick et al. (2009) met behulp van een waarde van 0,5 voor de scalaire c. om de kwaliteit van de DEBV te waarborgen, alleen dieren met een wegingsfactoren groter dan nul en een betrouwbaarheid van de DEBV groter dan 0.,20 werden gebruikt in de associatieanalyses. De betrouwbaarheid van de DEBV werd ook verkregen volgens Garrick et al. (2009).
Associatieanalyses werden uitgevoerd met behulp van de software ASREML (Gilmour et al.,, 2009) waarbij het volgende lineaire gemengde diermodel wordt toegepast:
waarbij DEBVij de waargenomen DEBV is voor dier j, w de wegingsfactor is voor het residu, µ het totale debv-gemiddelde van de populatie, Ri de draagstatus is (telling van het schadelijke allel) van de 4-mutatie i, aj het additieve genetische effect is geschat met behulp van een op stamboom gebaseerde gemiddelde relatiematrix, en de residufout . Associaties met A-log10 (p-waarde) groter dan vijf werden als significant verklaard.
resultaten
A 1.,5 Mb Segment op chromosoom 6 beïnvloedt lactatie overleving bij varkens
we analyseerden 31.638 dieren uit een enkele raszuivere varkenslijn (synthetische lijn met grote witte achtergrond), genotypeerd op de varkens 50K SNP chip (Sscrofa11.1 build) (Warr et al., 2019). De analyse toonde een 1.5 Mb segment op chromosoom 6 (SSC6:48.75–50.25) die een tekort in homozygositeit geassocieerd met verminderde lactatieoverleving tonen (tabellen 1 en 2). Het haplotype segregeert in de onderzochte populatie met een matige allelfrequentie van 4,5% (9,0% draagfrequentie)., De haplotypefrequentie schommelt de laatste tien jaar, maar is de laatste drie jaar afgenomen (figuur S1). We hebben getest of de frequentie werd aangedreven door een heterozygoot voordeel effect. Echter, we vonden meestal negatieve associaties met belangrijke selectiekenmerken behalve voor lendendiepte en drachtlengte (Tabel 3), wat suggereert dat de frequentie puur het gevolg is van genetische drift.

Tabel 1 haplotypekenmerken van SSC6.,

Tabel 2 draagdier-per-draagdier nesten vertonen een daling van 24% in de lactatieoverleving vergeleken met draagdier-per niet-draagdier nesten. Belangrijke resultaten worden vet gedrukt.

Tabel 3 kenmerken die significant geassocieerd zijn met heterozygote dragers van de sptbn4-deletie.
de 52 nesten per drager (CxC) vertonen geen significante afname van het totale aantal geboren of levende dieren., De lactatieoverleving is echter met ongeveer 24% verminderd bij CXC-nesten in vergelijking met draagdier-per-noncarrier (CxN) – paringen, wat erop wijst dat homozygote biggen sterven binnen de lactatieperiode (Tabel 2). Vervolgens onderzochten we de opmerkingen voor tijd en oorzaak van sterfte van CXC nesten. Hieruit bleek dat de meeste biggen die binnen de eerste 24 uur na de geboorte stierven. De meerderheid van deze biggen werden door boeren meestal beschreven als “zwak Biggetje bij de geboorte.,”
gehele Genoomsequentieanalyse toont een 16-bp Frameshift deletie aan in SPTBN4 als de waarschijnlijke causatieve Variant
om de causale mutatie te identificeren, hebben we gegevens over de gehele genoomsequentie van 71 dieren uit de onderzochte populatie onderzocht en vijf dragerdieren geïdentificeerd. De analyse van het verband tussen onevenwichtigheden (LD) toonde 267 SNP-en Indel-varianten in hoge LD (r2 > 0.8) aan met het haplotype SSC6 (tabel S1), De meerderheid in perfecte LD (247 varianten)., Slechts vijf varianten kunnen de codering beïnvloeden (drie missense, één frameshift, één splice-acceptor). De drie missense varianten worden naar verwachting getolereerd door SIFT (score > 0,18, tabel S1), terwijl de splice-acceptor variant een gen beïnvloedt dat codeert voor een 28 bp peptide met onbekende functie, waarschijnlijk niet Causaal. Echter, een variant in volledige LD (r2 = 1) met het haplotype werd voorspeld om een hoge impact te hebben; een 16-bp frameshift deletie in exon 26 van het sptbn4 Gen (6:g.48801280delgacggtgtacgcggt) (figuren 1A, B)., De frameshift deletie (Enssscp00000031537: P.Arg1902fs) introduceert 30 nieuwe aminozuren en een voortijdig stopcodon, waardoor een verminderd en afgeknot spectrin beta niet-erytrocytisch 4 eiwit (SPTBN4) ontstaat. Mutanten missen de uiteindelijke 662 aminozuren van het wild type eiwit (figuur 1C), met inbegrip van de pleckstrine homologie (PH) domein vereist voor eiwit transport naar membranen (Wang et al., 2018). De proteã ne SPTBN4 is een lid van de BÃ ta-spectrin proteã nen en is actin dat het celmembraan met actin cytoskeleton verbindt., Sptbn4 mutaties verstoren de cytoskeletale machines die de juiste lokalisatie van ionenkanalen in myelinated zenuwen regelen en motorische neuropathieën veroorzaken (Parkinson et al., 2001; Wang et al., 2018).

figuur 1 (A) sptbn4 genmodel. De locatie van de getroffen 26e exon is aangegeven in het rood. B) illustratie van de 16-bp-schrapping. Figuur toont wild type en mutant exon. C) uitlijning van de mutant (Mt) en het wildtype (Wt) SPTBN4-eiwitvolgorde. De mutatie induceert 30 nieuwe aminozuren en een voortijdig stopcodon.,
genotypering van vijf CXC-nesten bevestigt Sptbn4-deletie als de waarschijnlijke boosdoener
we hebben vijf CXC-nesten gegenotypeerd voor de 16-bp-deletie met ten minste twee biggen (bereik 2-6) die binnen de eerste 48 uur na de geboorte stierven. De vijf nesten produceerden 53 biggen waarvan 19 homozygoot waren voor de 16 bp deletie (Tabel 4). Alle 19 homozygote biggen stierven binnen 48 uur na de geboorte (18 binnen 24 uur). Van de 34 resterende biggen (8 wilde biggen en 26 dragers) stierven er slechts 1 binnen 48 uur, waarschijnlijk veroorzaakt door andere (milieu) factoren.,

Tabel 4 genotypering van de waarschijnlijke causale 16-bp SPTBN4 frameshift deletie in vijf draagdier-voor-draag nestjes. De som per genotype-klasse is vet gedrukt.
biggen homozygoot voor de Sptbn4-deletie lijden aan myopathie en verlamming van de achterste ledematen
we hebben één recent CXC-nest (werpdatum: 28 April 2019) gecontroleerd dat zes gezonde, twee aangetaste biggen (monsters: 9912, 9916) (figuur 2A) en drie doodgeboren biggen opleverde., We bevestigden de homozygote sptbn4 deletie status voor de twee getroffen biggen (tabel S2). Bovendien zagen we vier heterozygote dragers en twee homozygote wilde biggen onder de gezonde individuen. Een van de doodgeboren biggen (Monster: 9921) was ook homozygoot voor de verwijdering, terwijl de andere twee heterozygoot waren. De aangetaste biggen lijden aan extreme spierzwakte( figuren 2B, C), verlamming van de achterpoten en tremoren (S1 Video). Daardoor konden de biggen niet lopen of drinken.,
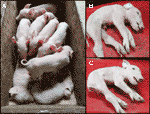
Figuur 2 (A) twee aangetaste biggen (levend) samen met zes gezonde nesten. De biggen zijn afkomstig van een CXC-dekking die op 28 April 2019 is gefokt. B) aangetaste Mannelijke Big 9912. C) aangetaste vrouwelijke Big 9916.
aangetaste biggen hebben geen Kruisstrepen in de skeletspieren van de rug en de achterste ledematen
histopathologisch onderzoek toonde verspreide degeneratie van spiervezels aan bij beide biggen, en focale necrose en vasculitis in de rugspier bij één van de biggen (ID = 9912)., Bovendien vertoont phosphotungstic acid hematoxyline (PTAH) kleuring divergente kleuren van de skeletspiervezels, wat wijst op afname van kruisstrepen, met name in de spieren van de dorsale en achterpoten van de getroffen dieren (figuur 3B), terwijl de voorpoten lijken onaangetast (figuur 3A). De afname van dwarsstrepen wordt aangegeven door abnormale kleuring en algemeen verlies in volume van spiervezels (figuur 3B). De histopathologisch waargenomen veranderingen in de achterpoten en in de dorsale spieren zijn indicatief voor spierdystrofie.,

Figuur 3 (A) dwarsdoorsnede van een skeletspier vanaf het voorbeen. De zwarte pijl geeft de normale kleur (donker) van spiervezels aan, wat wijst op de aanwezigheid van dwarsstrepen. PTAH Bar = 50 µm. B) dwarsdoorsnede van een skeletspier vanaf de achterpoot. De zwarte pijl geeft abnormale kleuring (roze) van spiervezels aangeeft gebrek aan kruisstrepen. De gele pijl geeft de normale kleur en de aanwezigheid van strepen aan. PTAH Bar = 50 µm.,
discussie
in dit onderzoek wordt melding gemaakt van een nieuw aangeboren defect dat biggensterfte veroorzaakt, waarschijnlijk als gevolg van een frameshift deletie van 16 bp in het sptbn4-gen. De biggen lijden aan extreme spierzwakte (myopathie) en sterven binnen enkele uren na de geboorte. De deletie zal naar verwachting leiden tot een volledig verlies van functie van het niet-erytrocytaire 4-eiwit spectrin bèta. SPTBN4 is een lid van de familie van spectrinegenen en voor ionenkanaal het clusteren bij de knopen van Ranvier vereist, beà nvloedend actiepotentieel (Devaux, 2010)., Mutaties verstoren de cytoskeletal machines die de juiste lokalisatie van ionenkanalen en functie van axonale domeinen controleren voornamelijk op de Axon initiële segmenten (AIS) en de knooppunten van Ranvier (Wang et al., 2018). Meer specifiek, het getroffen C-terminal domein van SPTBN4 is cruciaal voor kcnq2 kanaal trafficking en prikkelbaarheid op knooppunten van Ranvier (Devaux, 2010).
vervolgonderzoek bracht gevallen bij mensen en muizen aan het licht die erop wezen dat het daaropvolgende syndroom waarschijnlijk niet onmiddellijk dodelijk zou blijken te zijn, maar eerder ernstige myopathie zou veroorzaken., Aan de hand van gegevens over het SNP-genotype met gemiddelde dichtheid, die beschikbaar zijn voor alle dieren in de fokpopulatie (n = 31.839), konden dragers worden geïdentificeerd. Onder die dragers was een zeug die ongeveer halverwege de zwangerschap was op het moment van identificatie, verwekt door een zwijn dat ook drager was. De fokkerij werd op de hoogte gesteld om het nest bij de geboorte te documenteren. Het waargenomen fenotype van de aangetaste biggen (myopathie, verlamming van de achterste ledematen, tremoren) kwam volledig overeen met wat werd waargenomen bij humane patiënten met homozygote functieverlies of samengestelde heterozygote mutaties in het sptbn4-gen (OMIM: 606214)., Twee van de menselijke patiënten hebben verlies-van-functiemutaties binnen het pH-domein (Wang et al., 2018), die ondersteunen dat een verlies van het pH-domein bij varkens waarschijnlijk zou leiden tot een volledig verlies van de functie van het sptbn4-eiwit. Bij de mens leiden vergelijkbare mutaties tot ernstige aangeboren myopathie veroorzaakt door de afwezigheid van spier type I vezels, neuropathie en doofheid (Knierim et al., 2017; Wang et al., 2018). Wang et al. (2018) ook waargenomen motorische axonale neuropathie bij verschillende patiënten gekenmerkt door congenitale hypotonie, diepe zwakte, en verlies van diepe peesreflexen door vroege kinderjaren., Bovendien onthulden zenuwbiopten verminderde nodale Na + kanalen en geen nodale KCNQ2 K + kanalen, wat de moleculaire pathologie onthulde die zenuwsysteem dysfunctie veroorzaakte. Daarom concluderen we dat deze frameshift variant de waarschijnlijke causale mutatie is die leidt tot het waargenomen fenotype en depletie van het homozygote genotype in de populatie. Toekomstige studies zouden zich kunnen concentreren op het maken van een knockout in vivo van het sptbn4 gen in varken, om het syndroom en bijbehorende fenotype in meer detail te bestuderen.,
we hebben geen degeneratie van spiervezels in de voorpoten waargenomen, terwijl de rug-en achterpootspiervezels duidelijk aangetast waren. Deze observatie kan gedeeltelijk de verlamming van de achterpoot verklaren, terwijl de voorpoten niet worden aangetast. De discrepantie tussen voor-en achterpoten spiervezels is ook beschreven bij trillende muizen, waarbij sptbn4 verlies-off-functie mutaties motorische neuropathie, verlamming van de achterpoten, tremoren en centrale doofheid veroorzaken (Parkinson et al., 2001; Komada en Soriano, 2002). Parkinson et al., (2001) beschrijf verminderde zenuwgeleidingssnelheden in heupzenuwen van muizen met trillende allelen die de perifere achterpoot neuropathie veroorzaken. Expressie van SPTBN4 in muizen is beperkt tot de hersenen, het ruggenmerg, en ischias zenuwen en niet waargenomen in skeletspieren, dus deze ziekte is voornamelijk een neuronaal defect. Over het algemeen blijft onduidelijk welk mechanisme de afwezigheid van symptomen in de voorpoten veroorzaakt. Deze” natuurlijke knockout ” bij varkens kan een nuttige bron zijn om de menselijke ziekte te bestuderen, aangezien varkens gewoonlijk een beter model zijn om menselijke ziekte te bestuderen in vergelijking met knaagdiersoorten., Bovendien kan het gevolg van het verlies van de SPTBN4-functie in meer detail worden bestudeerd.
de effectieve populatiegrootte (Ne) van het onderzochte ras wordt geschat op ongeveer 100 (Hidalgo et al., 2016). Bij het fokken van dieren verhoogt lage Ne het risico dat schadelijke allelen bij toeval in frequentie toenemen. Bovendien hebben eerdere studies aangetoond dat recessieve letale allelen kunnen worden gedreven door voordelige effecten in heterozygoten (Derks et al., 2018; Matika et al., 2019). Matika et al.,, 2019 vond een recessieve stop-opgedaan mutatie in het mstn gen geassocieerd met een belangrijke toename van spierdiepte in heterozygoten. Echter, we vinden geen bewijs voor een heterozygote voordeel in onze studie. Met de huidige genomische technieken kunnen we nu schadelijke allelen identificeren die afdrijven naar hogere frequenties, en de opkomst van nieuwe schadelijke allelen nauwkeurig monitoren, waardoor meer effectieve purging mogelijk is. Bovendien zal het resultaat van dit soort onderzoek het bewustzijn van “verborgen” genetische defecten op zowel het niveau van de fokker als de Boer sterk verbeteren., Zonder voorafgaande informatie worden zeldzame aangeboren afwijkingen vaak geregistreerd als ” zwak Biggetje.”En zonder verder onderscheid van specifieke syndromen is verdere actie niet mogelijk. In de meeste gevallen is het Onbekend of er een genetische basis is, of dat er andere verstorende effecten kunnen zijn. Met voorafgaande genomic informatie, kan het syndroom worden geà dentificeerd, in vergelijking met andere gevallen, en die dragers worden geà dentificeerd, die tot actionable informatie leiden.
Biggensterfte is van groot economisch belang en van belang voor het dierenwelzijn., Daarom heeft de ontdekking van de mutatie van SPTBN4 geleid tot onmiddellijke implementatie in het fokprogramma om de frequentie van carrier-by-carrier paringen te minimaliseren. Dit maakt het mogelijk de geboorte van getroffen personen te voorkomen, waardoor het dierenwelzijn wordt verbeterd en de economische verliezen worden verminderd.
conclusie
in deze studie rapporteren we een nieuw aangeboren defect dat waarschijnlijk wordt veroorzaakt door een recessieve frameshift deletie in het sptbn4 gen bij varkens. De bevindingen worden ondersteund door opvallende overeenkomsten met sptbn4 geassocieerde Syndrome fenotypen bij mensen en muizen., De studie maakt het mogelijk om het schadelijke allel van de bevolking te controleren en te zuiveren. Kruisingen tussen vervoerders kunnen worden voorkomen, waardoor getroffen personen worden uitgesloten, waardoor economische verliezen worden beperkt en het dierenwelzijn wordt verbeterd. Ten slotte kunnen deze” natuurlijke knockouts ” verkregen in de fokindustrie een model voor menselijke ziekte bieden en ons begrip van genfunctie binnen de zoogdierklade vergroten, en een potentieel model voor menselijke ziekte bieden.,
Data Availability Statement
Ethics Statement
ethische beoordeling en goedkeuring was niet vereist voor het dieronderzoek, omdat de in dit onderzoek gebruikte gegevens zijn verkregen als onderdeel van routinematige gegevensverzameling van Topigs Norsvin fokprogramma ‘ s, en niet specifiek voor dit project. Daarom was de goedkeuring van een ethische commissie niet verplicht. Monsterverzameling en gegevensregistratie werden strikt uitgevoerd volgens de Gezondheids – en welzijnswet voor dieren., Van de eigenaren werd schriftelijke geïnformeerde toestemming verkregen voor de deelname van hun dieren aan dit onderzoek.
Auteursbijdragen
MG, H-JM en MD ontwierpen en ontwierpen het onderzoek. BH was verantwoordelijk voor de algemene organisatie en communicatie met Topigs Norsvin en boeren. MD en ML hebben de gegevensanalyse uitgevoerd. BD en KL voerden laboratoriumwerk uit. SG-V heeft de pathologische analyse uitgevoerd. MD schreef het manuscript. H-JM, MG, BH, SG-V, BD, KL en ML gaven nuttige opmerkingen en suggesties en hielpen bij het opstellen van het manuscript. Fenotypische gegevens werden geanalyseerd per ML., Alle auteurs hebben het definitieve manuscript gelezen en goedgekeurd.
financiering
Dit onderzoek werd gefinancierd door het STW-Breed4Food Partnership, projectnummer 14283: From sequence to fenotype: detecting deleterious variation by prediction of functionality. Dit onderzoek werd financieel ondersteund door NWO-TTW en de Breed4Food partners Cobb Europe, CRV, Hendrix Genetics en Topigs Norsvin. Daarnaast werd deze studie ondersteund door het IMAGE project (Horizon 2020, Nr. 677353). De financiers hadden geen rol in het ontwerp van de studie, het verzamelen en analyseren van gegevens, het besluit tot publicatie of de voorbereiding van het manuscript., Het gebruik van de HPC cluster werd mogelijk gemaakt door CATAgroFood (Shared Research Facilities Wageningen UR).
Disclaimer
De in dit onderzoek gebruikte gegevens zijn verkregen als onderdeel van routinematige gegevensverzameling van fokprogramma ‘ s van Topigs Norsvin, en niet specifiek voor dit project. Daarom was de goedkeuring van een ethische commissie niet verplicht. Monsterverzameling en gegevensregistratie werden strikt uitgevoerd volgens de Gezondheids – en welzijnswet voor dieren.,
belangenconflict
ML en BH zijn werknemers van Topigs Norsvin Research Center, een onderzoeksinstituut dat nauw verwant is aan een van de financiers (Topigs Norsvin).
alle overige auteurs verklaren dat het onderzoek werd uitgevoerd zonder enige commerciële of financiële relatie die als een potentieel belangenconflict zou kunnen worden opgevat.de andere Breed4Food partners Cobb Europe, CRV, Hendrix Genetics, verklaren geen concurrerende belangen te hebben voor deze studie.,Dankbetuigingen de auteurs danken Frank Van Haaren en Toon Janssen voor het organiseren van de communicatie met de boerderij. We danken Manon Houben voor haar bijdrage aan de pathologische onderzoeken. Wij danken Mout Viller voor het maken van video ’s en foto’ s van de getroffen en gezonde biggen na de geboorte. Wij danken Jürgen Harlizius voor het verzorgen van de biggen na de overdracht. Wij danken Gerda Van Eldik voor de monsterverzameling. Wij danken Egbert Knol en Egiel Hanenberg voor de algemene steun en begeleiding van Topigs-Norsvin.,
aanvullend materiaal
Video S1 / Video toont beide getroffen personen na de geboorte.
tabel S1 / genomische variatie in hoge LD met het haplotype SSC6.
tabel S2 / genotypering van het CXC-nest met twee getroffen individuen (kraamdatum: 28 April 2019). Tabel toont homozygote del / del status voor de twee getroffen biggen 9912,9916.
tabel S3 / Mapping en dekkingsstatistieken van WGS-monsters.
figuur S1 / SPTBN4 deletiedragerfrequentie vanaf 2007-2019.