Introdução
Leitão de mortalidade é um dos principais seleção de traços em criações de suínos, e é influenciado pelo semear, leitões, e o meio ambiente. Portanto, a mortalidade dos leitões é um fenótipo complexo e depende da capacidade da porca para criar seus descendentes, mas também é uma função do peso do nascimento, manejo e seleção (Knol et al., 2002)., No entanto, também os defeitos recessivos monogênicos contribuem para a mortalidade dos Leitões, embora apenas alguns exemplos tenham sido relatados no passado (Murgiano et al., 2012; Matika et al., 2019). Mesmo nos casos em que o efeito da mutação é grave, a selecção eficiente contra essa mutação é dificultada pela baixa frequência. Em muitos defeitos graves, os zigotos morrem muito cedo na gestação, não deixando nenhum traço a não ser a ausência de homozigotos na população em geral (Derks et al., 2019).,os efeitos de consanguinidade nas populações de suínos comerciais são geralmente controlados através da criação selectiva de uma diminuição da mortalidade nos leitões, melhorando tanto as capacidades de mãe como a viabilidade dos Leitões (Olijslagers, 2018). No entanto, as variantes subjacentes aos defeitos monogênicos recessivos não são bem capturadas dentro dos valores de reprodução, e potencialmente derivam para frequências mais altas como resultado de uma seleção intensa (Georges et al., 2019). Além disso, essas variantes também podem ser mantidas como resultado da seleção de equilíbrio para um efeito positivo correlacionado no estado heterozigótico (Derks et al., 2018).,os defeitos recessivos apenas contribuem marginalmente para a mortalidade global dos Leitões (Alonso-Spilsbury et al., 2007). No entanto, as variantes que afectam a mortalidade dos leitões são de grande importância porque influenciam directamente a produção e o bem-estar dos animais (Baxter et al., 2013; Rutherford et al., 2013). No entanto, na gestão da população animal, a baixa frequência de ocorrência de defeitos é geralmente mal documentada (muitas vezes, termos muito gerais são usados), e síndromes são muitas vezes reconhecidos apenas uma vez que eles tenham atingido uma alta frequência., Isto é especialmente relevante para síndromes que não levam a fenótipos muito distintos. Portanto, mesmo em populações de reprodução comercial pouco rastreamento pode ser feito em síndromes específicas, e para efetivamente selecionar contra síndromes específicas de baixa frequência, portanto, requer novas abordagens.
neste trabalho, descrevemos a descoberta de uma síndrome altamente debilitante em uma população de porcos comerciais através de um levantamento baseado em matrizes combinadas de média densidade SNP e sequenciamento de genoma inteiro (WGS)., A pesquisa levou à identificação de uma deleção frameshift de 16-bp no gene SPTBN4, com consequências fenotípicas claras previstas em homozigotos. A frequência portadora é de cerca de 9% na população em estudo, afetando aproximadamente 0,81% das ninhadas da população. A frequência era suficientemente baixa para ser desconhecida para ter uma base genética, e mesmo efetivamente não reconhecida como uma síndrome específica. Após a implementação da pesquisa, uma porca grávida foi identificada por um javali., Os leitões afetados sofrem de miopatia e são incapazes de andar, geralmente resultando em morte em poucas horas após o nascimento, completamente de acordo com a patologia prevista em comparação com casos semelhantes de humanos e ratos.Material e métodos animais, genótipos e pré-processamento o conjunto de dados é constituído por 31 839 animais de uma linha sintética de javalis de fundo branco grande. A linha é mantida e criada em Topigs Norsvin nucleus farms, principalmente selecionando características de produção e saúde., Os animais foram genotipados no Illumina GeneSeek custom 50K SNP chip (Lincoln, NE, EUA). Os animais com uma frequência de genótipos em falta > 0.15 foram removidos. Nós descartados marcadores que não satisfazem os seguintes critérios de filtragem: Um mínimo de chamada taxa de 0,85, um menor alelo frequência > 0.01, e um de Hardy-Weinberg proporções teste exato de p-valor abaixo de P < 10-12. Além disso, marcadores com localização desconhecida na construção do genoma Sscrofa11.1 foram descartados, deixando 41.573 marcadores após filtragem., Todas as etapas foram realizadas em Plink v1. 90b3. 30 (Purcell et al., 2007).
Haplotype Phasing and Identification of SSC6 Haplotype
realizámos haplotype phasing and imputation of missing sites in Beagle5. 0 with parameter for effective population size set to 100, other settings were default (Browning et al., 2018). As homozigotas esperadas foram estimadas com base na frequência de haplotipos, usando o princípio Hardy-Weinberg. Um teste binomial exato foi aplicado para testar o número de homozigotos observados com o número de homozigotos esperados., O haplótipo foi considerado significativamente depauperado se P < 5 × 10-3.
Efeitos Fenotípicos Associados Com SSC6 Haplótipo
Nós examinamos a SSC6 de haplótipos para os registos no número total de nascidos, número de natimortos, mumificados leitões, farrowing sobrevivência, e lactação sobrevivência (sobrevivência até cerca de 21 dias de idade), para um total de 9,666 ninhadas. Listamos estes fenótipos para todas as ninhadas CxC e CXN identificadas. Usamos um teste T de Welch para avaliar se os fenótipos das ninhadas CxC diferem significativamente das ninhadas CxN. A p-value < 0.,05 foi considerado significativo.
análise da sequenciação do genoma total e Identificação da variante candidata
o conjunto de dados consiste em 71 indivíduos sequenciados do genoma inteiro da população em estudo. Todos os 71 amostras foram também presente em nosso conjunto de dados de 31,839 animais genotipada no 50K. 71 amostras têm um volume total de 1.93 Tbp (tera de pares de base) a partir de 14.16 bilhões de 150 bp emparelhado-end lê (Tabela S3). As amostras foram sequenciadas em Illumina HiSeq 2000. Alinhamos as sequências com a construção do genoma Sscrofa11. 1 usando a versão 0.7 do BWA-MEM.,15 (Li e Durbin, 2009) com uma mapeabilidade média de 98,9% e uma cobertura de amostra variando de 8,8 a 14,8 X (10,9 X média). Samblaster foi usado para remover duplicados PCR (Faust e Hall, 2014). O Samtools foi usado para ordenar, fundir e indexar arquivos bam (Li et al., 2009). O mapeamento e as estatísticas de qualidade foram gerados utilizando o Qualimap (Okonechnikov et al., 2016). Variante chamada foi realizada com Freebayes v1.1.0 com as seguintes configurações: –min-base-Qualidade 10 –min-alternate-fraction 0.2 –haplotype-length 0 –min-alternate-count 2 (Garrison e Marth, 2012)., Variantes com phred quality score < 20 foram descartadas (Li et al., 2009). Variants were annotated using the Ensembl variant effect predictor (VEP, release 96) (Mclaren et al., 2016). O impacto das variantes de missense foi previsto usando a classificação intolerante da tolerante (SIFT) (Kumar et al., 2009). A análise LD foi realizada utilizando Plink v1. 90b3. 30 (Purcell et al., 2007) com as seguintes configurações –chr-set 18, –r2, ld-window-r2 0.8.o alinhamento proteico entre o tipo selvagem e a proteína mutante foi realizado utilizando ClustalO (Madeira et al.,, 2019) e visualizado usando ESPript 3 (Robert e Gouet, 2014). Mais Visualização e validação foram realizadas usando a versão 1.12.1 do genoma JBrowse (Skinner et al., 2009).
Validação de Causalidade 16 bp SPTBN4 de Eliminação
PCR foi realizada com 60 ng de DNA genômico, com 0,4 µm de cada primer, de 1,8 mM MgCl2, e de 25 unidades/ml OneTaq® DNA Polymerase (OneTaq® 2X Master Mix com Padrão de Buffer, New England Biolabs) na fabricante de PCR buffer em um volume final de 12 µl., A desnaturação inicial de 1 minuto a 95 ° C foi seguida por 35 ciclos de 95°C para 30 s, 55°C para 45 s, 72°C 90 s, seguido por uma extensão de 5 min 72°C. Os primadores de PCR para SPTBN4 são TCAAGGGTGCAGGCTCTTC para a frente e GGTAGGCTCGAAGTGGG reverso. O primário dianteiro foi marcado com 6-FAM para produzir um produto PCR fluorescentemente marcado detectável em ABI 3730 DNA sequencer (Applied Biosystems). Os tamanhos de fragmentos foram determinados usando o GeneMapper software 5 da ABI.,dois leitões afectados com menos de uma semana de idade foram enviados para exame ao departamento de patologia da Real Saúde Animal (Deventer). Macroscopicamente, todas as observações estavam dentro dos limites normais. O músculo esquelético da perna dianteira, o músculo dorsal e a parte de trás de ambos os animais foram amostrados para a rotina H&e coloração e PTAH. O tecido muscular foi armazenado em frascos separados e fixado em solução de formaldeído a 4%, tamponado (=solução de formalina a 10%, tamponado)., Depois disso, o tecido foi incorporado em parafina e cortado em 2 µm de acordo com o procedimento de operação padrão (SOP RAH). Posteriormente, os diapositivos foram desparafinizados e rotineiramente manchados para hematoxilina e eosina (H&e) em uma máquina de cor automática. Simultaneamente, lâminas adicionais de 2 µm do tecido muscular, bem como uma lâmina de controle positivo do tecido muscular foram preparados para a coloração manual com “hematoxilina ácido fosfotungstico”, abreviado como PTAH. Esta coloração é preferida para demonstrar estrias cruzadas do músculo esquelético.,neste estudo, avaliamos 63 traços utilizados no programa de reprodução. Valores estimados de reprodução desregulamentados (DEBV) foram utilizados como uma variável de resposta para cada característica em estudo. O valor estimado de reprodução (EBV) de todos os traços avaliados foi desregulamentado usando a metodologia descrita por Garrick et al. (2009). O EBV de cada animal foi obtido a partir da avaliação genética de rotina por um programa comercial de reprodução (Topigs Norsvin) usando um modelo animal., As confiabilidades por animal para fins de desregulamentação foram extraídas da avaliação genética baseada na metodologia de Tier e Meyer (2004). As heritabilidades usadas para a desregulação também foram extraídas da avaliação genética de rotina. Por último, os factores de ponderação baseados na fiabilidade estimada do DEBV foram também estimados de acordo com Garrick et al. (2009) utilizando um valor de 0,5 para o escalar C. para garantir a qualidade do DEBV, apenas animais com factores de ponderação superiores a zero e uma fiabilidade do DEBV superior a 0. ,20 foram utilizados nas análises da Associação. A fiabilidade do DEBV também foi obtida de acordo com Garrick et al. (2009).as análises de associação foram realizadas usando o software ASREML (Gilmour et al.,, 2009) a aplicação da seguinte linear misto modelo animal:
onde DEBVij é o observado DEBV para o animal j, w é o fator de ponderação para o residual, µ é o DEBV média da população, o Ri é o estado de portador (contagem da prejudicial de alelos) de 4 de mutação i, aj é o aditivo genética efeito estimado através de uma árvore genealógica baseada em média, matriz de relacionamento, e o erro residual. As associações com a-log10 (valor P) superior a cinco foram declaradas como significativas.
resultados
a 1.,5 Mb Segment on Chromosome 6 Affects Lactation Survival in Pigs
we analyzed 31. 638 animals from a single purebred boar line( synthetic line with large white background), genotiped on the Porcine 50K SNP chip (Sscrofa11.1 build) (Warr et al., 2019). A análise revelou um segmento de 1,5 Mb no cromossoma 6 (SSC6:48,75–50,25) mostrando um déficit na homozigosidade associada à redução da sobrevivência da lactação (Tabelas 1 e 2). O haplótipo é segregado com uma frequência alélica moderada de 4, 5% (9, 0% de Frequência portadora) na população em estudo., A frequência de haplotipos tem flutuado ao longo da última década, mas diminuiu ao longo dos últimos 3 anos (figura S1). Nós testamos se a frequência foi impulsionada por um efeito de vantagem heterozigótica. No entanto, encontramos principalmente associações negativas com traços de seleção importantes, exceto para a profundidade do lombo e comprimento da gestação (Tabela 3), o que sugere que a frequência é puramente o resultado da deriva genética.

Tabela 1 SSC6 haplótipo características.,

Tabela 2 as ninhadas portadoras-portadoras apresentam uma diminuição de 24% na sobrevivência da lactação em comparação com as ninhadas não portadoras. Os resultados significativos são indicados a negrito.

Tabela 3 Características significativamente associados com os portadores heterozigóticos da SPTBN4 exclusão.
as 52 ninhadas transportadoras (CxC) não apresentam uma redução significativa do número total de animais nascidos ou nascidos em livre prática., No entanto, a sobrevivência à lactação é reduzida em cerca de 24% nas ninhadas CxC comparativamente às matagens carrier-by-noncarrier (CxN), indicando que os leitões homozigóticos morrem durante o período de lactação (Tabela 2). Em seguida, examinamos as observações por tempo e causa da mortalidade das ninhadas CxC. Isto revelou que a maioria dos leitões que morreram nas primeiras 24 horas após o nascimento. A maioria desses leitões foram descritos principalmente pelos agricultores como “leitões fracos à nascença.,”
A análise da sequenciação do genoma total revela uma eliminação de 16 bp em Frameshift no SPTBN4 como a variante causativa provável
para identificar a mutação causal, examinámos os dados da sequência do genoma total de 71 animais da população em estudo e identificámos cinco animais portadores. Linkage disequilibrium (LD) analysis revealed 267 SNP and indel variants in high LD (r2 > 0.8) with the SSC6 haplotype (Table S1), the majority being in perfect LD (247 variants)., Apenas cinco variantes potencialmente afetam a sequência de codificação (três missense, um frameshift, um aceitador de splice). Prevê-se que as três variantes do missense sejam toleradas pelo SIFT (score > 0.18, tabela S1), enquanto que a variante do splice-acceptor afeta um gene que codifica um peptídeo de 28 bp de função desconhecida, improvável que seja causal. No entanto, uma variante em LD completa (r2 = 1) com o haplótipo foi prevista para ter alto impacto; uma deleção de frameshift de 16-bp em exon 26 do gene SPTBN4 (6:g. 48801280delgacgggtgtacgcggt) (figuras 1A, B)., A deleção frameshift (ENSSSCP00000031537:P. Arg1902fs) introduz 30 aminoácidos novos e um codão de paragem prematura, produzindo uma proteína beta não eritrocítica 4 deficiente e truncada (SPTBN4). Os mutantes não possuem os 662 aminoácidos finais da proteína de tipo selvagem( figura 1C), incluindo o domínio de homologia da pleckstrina (PH) necessário para o transporte de proteínas para membranas (Wang et al., 2018). A proteína SPTBN4 é um membro das proteínas beta-espectrinas e é uma actina que liga a membrana celular ao citoesqueleto de actina., As mutações SPTBN4 perturbam a maquinaria do citoesqueleto que controla a localização adequada dos canais iónicos nos nervos mielinizados que causam neuropatias motoras (Parkinson e outros)., 2001; Wang et al., 2018).

Figura 1 (a) modelo genético SPTBN4. A localização do 26º exon afectado é indicada em vermelho. B) ilustração da supressão de 16-bp. A figura mostra o tipo selvagem e exon mutante. C) alinhamento da sequência proteica SPTBN4 mutante (Mt) e selvagem (Wt). A mutação induz 30 novos aminoácidos e um codon de paragem prematura.,
a genotipagem de cinco ninhadas CxC confirma a eliminação de SPTBN4 como o provável culpado
nós genotipámos cinco ninhadas CxC para a eliminação de 16-bp que tinham pelo menos dois leitões (intervalo 2-6) que morreram nas primeiras 48 horas após o nascimento. As cinco ninhadas produziram 53 leitões, dos quais 19 eram homozigóticos para a supressão de 16 bp (Tabela 4). Todos os 19 leitões homozigóticos morreram em 48 horas após o nascimento (18 em 24 horas). Dos 34 leitões restantes (8 tipos selvagens e 26 portadores), apenas 1 morreu dentro de 48 horas, provavelmente causada por outros fatores (ambientais).,

Tabela 4 genotipagem da provável supressão causal de 16-bp SPTBN4 frameshift em cinco liteiras portadoras-portadoras. A soma por classe de genótipo é indicada a negrito.
Leitões Homozigotos para o SPTBN4 Exclusão Sofrem De Miopatia e Posteriores Paralisia
Nós monitorados recente CxC maca (farrowing data: 28 de abril de 2019), que produziu seis saudável, dois afetado (exemplos: 9912, 9916) (Figura 2A), e três leitões natimortos., Confirmamos o estado de exclusão homozigótica do SPTBN4 para os dois leitões afetados (tabela S2). Além disso, observamos quatro portadores heterozigóticos e dois leitões homozigóticos selvagens entre os indivíduos saudáveis. Um dos Leitões natimortos (amostra: 9921) também foi homozigótico para a eliminação, enquanto os outros dois eram heterozigóticos. Os leitões afectados sofrem de fraqueza muscular extrema (figuras 2B, c), paralisia dos membros traseiros e tremores (vídeo S1). Por isso, os leitões não podiam andar nem beber.,
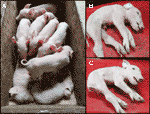
Figura 2 a) dois leitões afectados (vivos) juntamente com seis ninhadas saudáveis. Os leitões derivam de um acasalamento CxC farrowed em 28 de abril de 2019. B) Leitão macho afectado 9912. C) Leitão fêmea afectada 9916.o exame histopatológico revelou degeneração dispersa das fibras musculares em ambos os leitões, e necrose focal e vasculite no músculo dorsal de um dos Leitões (ID = 9912)., Além disso, a coloração do ácido fosfotúngstico hematoxilina (PTAH) mostra coloração divergente das fibras do músculo esquelético, indicando diminuição das estrias cruzadas, particularmente nos músculos das pernas dorsal e traseira dos animais afetados (figura 3B), enquanto as pernas dianteiras parecem não afetadas (figura 3A). A diminuição das estrias cruzadas é indicada por coloração anormal e perda geral de volume de fibras musculares (figura 3B). As alterações histopatologicamente observadas nas patas traseiras e nos músculos dorsais são indicativas de distrofia muscular.,

a Figura 3 (A) visão Transversal de um músculo esquelético da perna da frente. A seta preta indica coloração normal (escura) das fibras musculares indicando presença de estrias cruzadas. PTAH Bar = 50 µm. B) visão transversal de um músculo esquelético da perna traseira. A seta preta indica coloração anormal (rosa) das fibras musculares indicando falta de estrias cruzadas. A seta amarela indica coloração normal e presença de estrias cruzadas. PTAH Bar = 50 µm.,
Discussão
neste trabalho relatamos um romance defeito congênito, causando mortalidade de leitões, provavelmente, devido a um 16 bp frameshift exclusão no SPTBN4 gene. Os leitões sofrem de extrema fraqueza muscular (miopatia) e morrem poucas horas após o nascimento. Espera-se que a deleção confira uma perda completa de função da espectrina beta, proteína 4 não-eritrocítica. O SPTBN4 é um membro da família de genes espectrônicos e é necessário para o agrupamento de canais iônicos nos nós de Ranvier, afetando o potencial de ação (Devaux, 2010)., As mutações perturbam a maquinaria do citoesqueleto que controla a localização adequada dos canais iónicos e a função dos domínios axonais, principalmente nos segmentos axon iniciais (AIS) e nos nós de Ranvier (Wang et al., 2018). Mais especificamente, o domínio C-terminal afetado do SPTBN4 é crucial para o tráfico do canal KCNQ2 e excitabilidade em nós de Ranvier (Devaux, 2010).pesquisas posteriores identificaram casos humanos e de ratinhos que indicavam que a síndrome que se seguiu provavelmente não seria imediatamente letal, mas conferia miopatia grave., Com base nos dados do genótipo SNP de média densidade, disponíveis para todos os animais da população reprodutora (N = 31.839), os portadores puderam ser identificados. Entre esses portadores estava uma porca que estava aproximadamente a meio da gravidez no momento da identificação, criada por um javali que também era portador. A exploração de reprodução foi notificada para documentar a ninhada à nascença. O fenótipo observado dos Leitões afetados (miopatia, paralisia dos membros traseiros, tremores) foi completamente congruente com o que foi observado em doentes humanos com perda homozigótica de função ou mutações heterozigóticas compostas no gene SPTBN4 (OMIM: 606214)., Dois dos pacientes humanos têm mutações de perda de funções dentro do domínio PH (Wang et al., 2018), sustentando que uma perda do domínio do PH nos suínos conduziria provavelmente a uma perda completa da função da proteína SPTBN4. Em humanos, mutações semelhantes levam a miopatia congênita grave causada pela ausência de fibras musculares tipo I, neuropatia e surdez (Knierim et al., 2017; Wang et al., 2018). Wang et al. (2018) também observou neuropatia motora axonal em vários doentes caracterizados por hipotonia congénita, fraqueza profunda e perda de reflexos dos tendões profundos na infância., Além disso, as biópsias nervosas revelaram canais na+ Nadal reduzidos e canais KCNQ2 K+ nodal, revelando a patologia molecular causando disfunção do sistema nervoso. Portanto, concluímos que esta variante frameshift é a provável mutação causal que leva ao fenótipo observado e depleção do genótipo homozigótico na população. Estudos futuros poderiam focar em fazer um nocaute in vivo do gene SPTBN4 no porco, para estudar a síndrome e fenótipo associado em mais detalhes.,
não observamos degeneração das fibras musculares nas pernas dianteiras, enquanto as fibras musculares dorsais e traseiras foram claramente afetadas. Esta observação pode explicar parcialmente a paralisia dos membros traseiros, enquanto as pernas dianteiras não são afetadas. A discrepância entre as fibras musculares das pernas dianteiras e traseiras também tem sido descrita em camundongos, em que as mutações de perda da função SPTBN4 causam neuropatia motora, paralisia dos membros traseiros, tremores e surdez central (Parkinson et al., 2001; Komada and Soriano, 2002). Parkinson et al., (2001) describe reduced nerve-conduction velocities in sciatic nerves of mice with quivering alleles causing the peripheral hind limb neuropatia. A expressão do SPTBN4 em ratos é restrita ao cérebro, medula espinhal e nervos ciáticos e não observado no músculo esquelético, então esta doença é principalmente um defeito neuronal. No geral, permanece incerto qual o mecanismo que causa a ausência de sintomas nos membros dianteiros. Este” knockout natural ” em porcos pode ser um recurso útil para estudar a doença humana, já que os porcos são geralmente um melhor modelo para estudar a doença humana em comparação com as espécies de roedores., Além disso, as consequências da perda da função SPTBN4 podem ser estudadas mais pormenorizadamente.o tamanho efectivo da população (Ne) da raça em estudo é estimado em cerca de 100 (Hidalgo et al., 2016). Na reprodução animal, o baixo nível de azoto aumenta o risco de aumento casual da frequência dos alelos prejudiciais. Além disso, estudos anteriores demonstraram que alelos letais recessivos podem ser impulsionados por efeitos vantajosos em heterozigotos (Derks et al., 2018; Matika et al., 2019). Matika et al.,, 2019 encontrou uma mutação recessiva no gene MSTN associada a um grande aumento na profundidade muscular em heterozigotos. No entanto, não encontramos nenhuma evidência para qualquer vantagem heterozigótica em nosso estudo. Com as técnicas genômicas atuais, podemos agora identificar alelos deletérios à deriva para frequências mais altas, e monitorar o surgimento de alelos deletérios novos com precisão, permitindo purga mais eficaz. Além disso, o resultado deste tipo de estudo melhorará grandemente a consciência dos defeitos genéticos “ocultos”, tanto a nível de criador como de agricultor., Sem qualquer informação prévia, defeitos de nascença raros são muitas vezes registrados como “piglet fraco.”E sem qualquer outra distinção de síndromes específicas, não é possível mais ação. Na maioria dos casos, desconhece-se se existe uma base genética ou se podem existir outros efeitos de confusão. Com informações genômicas anteriores, a síndrome pode ser identificada, em comparação com outros casos, e portadores identificados, levando à informação acionável.a mortalidade dos leitões é de elevada importância económica e para o bem-estar dos animais., Assim, a descoberta da mutação SPTBN4 levou à implementação imediata no programa de criação para minimizar a frequência de matagens porta-a-porta. Isto permite evitar o nascimento de indivíduos afectados, melhorando assim o bem-estar dos animais e reduzindo as perdas económicas.
conclusão
neste estudo, reportamos um novo defeito congénito, provavelmente causado por uma deleção recessiva da transformação no gene SPTBN4 em suínos. Os achados são apoiados por semelhanças marcantes com os fenótipos sindromáticos associados ao SPTBN4 em humanos e ratos., O estudo permite monitorizar e eliminar o alelo deletério da população. Os cruzamentos porta-a-porta podem ser evitados, impedindo os indivíduos afectados, reduzindo assim as perdas económicas e melhorando o bem-estar dos animais. Finalmente, estes “knockouts naturais” obtidos na indústria de reprodução podem fornecer um modelo para a doença humana e aumentar a nossa compreensão da função genética dentro do clado mamífero, e fornecer um modelo potencial para a doença humana.,
Dados de Disponibilidade Declaração
Ética Instrução
análise e aprovação Ética não era necessário para o estudo em animais, porque os dados utilizados neste estudo foram obtidos como parte da rotina de coleta de dados a partir de Topigs Norsvin programas de melhoramento genético, e não especificamente para o propósito deste projeto. Por conseguinte, a aprovação de um comité de ética não era obrigatória. A recolha de amostras e o registo de dados foram realizados estritamente de acordo com a lei neerlandesa sobre protecção e bem – estar dos animais (gezondheid-en welzijnswet voor dieren)., Os proprietários deram o seu consentimento por escrito para a participação dos seus animais neste estudo.
contribuições dos autores
MG, H-JM, E MD conceberam e conceberam o estudo. A BH era responsável pela organização geral e comunicação com os Topigs Norsvin e os agricultores. MD e ML realizaram a análise de dados. BD e KL realizaram trabalhos de laboratório. A SG-V realizou a análise patológica. MD escreveu o manuscrito. H-JM, MG, BH, SG-V, BD, KL e ML forneceram comentários e sugestões úteis e ajudaram a redigir o manuscrito. Os dados fenotípicos foram analisados por ML., Todos os autores lêem e aprovam o manuscrito final.
financiamento
esta investigação foi financiada pela parceria STW-Breed4Food, projecto número 14283: da sequência ao fenótipo: detecção de variações deletérias por previsão da funcionalidade. Este estudo foi apoiado financeiramente pela NWO-TTW e pelos Breed4Food partners Cobb Europe, CRV, Hendrix Genetics e Topigs Norsvin. Além disso, este estudo foi apoiado pelo projeto IMAGE (Horizonte 2020, n. º 677353). Os financiadores não tiveram papel no desenho do estudo, na coleta e análise de dados, na decisão de publicar ou na preparação do manuscrito., A utilização do cluster HPC foi possível graças à CATAgroFood (instalações de investigação partilhadas de Wageningen UR).
Declaração de exoneração de Responsabilidade
os dados utilizados neste estudo foram obtidos como parte da recolha de dados de rotina de Programas de reprodução de Topigs Norsvin, e não especificamente para o propósito deste projeto. Por conseguinte, a aprovação de um comité de ética não era obrigatória. A recolha de amostras e o registo de dados foram realizados estritamente de acordo com a lei neerlandesa sobre protecção e bem – estar dos animais (gezondheid-en welzijnswet voor dieren).,
Conflito de Interesse
ML e BH são funcionários da Topigs Norsvin Research Center, um instituto de pesquisa estreitamente relacionado com um dos financiadores (Topigs Norsvin).todos os autores restantes declaram que a pesquisa foi realizada na ausência de quaisquer relações comerciais ou financeiras que possam ser interpretadas como um potencial conflito de interesses.os outros Parceiros do Breed4Food Cobb Europe, CRV, Hendrix Genetics, declaram não ter interesses concorrentes para este estudo.,
agradecimentos
os autores agradecem Frank van Haaren e Toon Janssen por organizarem a comunicação com a fazenda. Agradecemos a Manon Houben pela sua contribuição para os exames patológicos. Agradecemos a Mout Viller por tirar vídeos e fotos dos Leitões afetados e saudáveis após o nascimento. Agradecemos a Jürgen Harlizius por cuidar dos Leitões depois da entrega. Agradecemos a Gerda van Eldik pela recolha de amostras. Agradecemos Egbert Knol e Egiel Hanenberg pelo apoio geral e orientação de Topigs-Norsvin.,
material suplementar
Vídeo S1 | vídeo mostrando ambos os indivíduos afetados após o nascimento.
Table S1 / Genomic variation in high LD with the SSC6 haplotype.tabela S2 / genotipagem da ninhada CxC, incluindo dois indivíduos afetados (data de parição: 28 de abril de 2019). A tabela mostra o estado homozigótico del/del para os dois leitões afetados 9912,9916.
quadro S3 / estatísticas de mapeamento e cobertura de amostras WGS.
figura S1 / SPTBN4 frequência portadora de supressão de 2007-2019.