Introduction
les staphylocoques à Coagulase négative (CoNS), membres du groupe des staphylocoques, sont caractérisés comme des cocci Gram-positifs, présentés comme des cellules uniques ou à disposition irrégulière, et sont immobiles, Non sporogènes, anaérobies, et manque de l’enzyme coagulase.,1 les inconvénients font partie du microbiote humain, considéré comme des pathogènes opportunistes, causant des infections principalement chez les bébés prématurés, et les patients immunodéprimés et prothétiques.2
la montée de la résistance aux antimicrobiens ces dernières années a eu un grand impact sur les infections hospitalières causées par les CoNS. L’oxacilline est une pénicilline semi-synthétique utilisée dans le test de sensibilité pour la détection de la résistance à la méthicilline et le traitement des infections staphylococciques. Cependant, des taux de résistance à l’oxacilline de 66% à 95% ont été observés dans les isolats cliniques CoNS.,2 La résistance à L’oxacilline est souvent médiée par le gène mecA, qui code une protéine supplémentaire de liaison à la pénicilline (PBP2a) de faible affinité pour les pénicillines semi-synthétiques.3 le gène mecA est situé sur un élément génétique mobile connu sous le nom de Chromosome Cassette staphylococcique mec (SCCmec) qui contient le complexe mec, composé du gène mecA et de ses gènes régulateurs mecI et mecRI, le complexe ccr, responsable de l’intégration et de l’excision du SCCmec, et la région J, qui n’est pas essentielle à la formation du SCCmec, mais peut porter des gènes de résistance non β-lactame.,4 à ce jour, 13 types de Cemcc ont été décrits, basés sur la combinaison de types de complexes géniques ccr et de classes de complexes géniques mec. Les sous-types ont été définis par des polymorphismes de la région J dans la même combinaison de complexes mec et ccr.5
L’émergence d’isolats résistants à l’oxacilline a conduit à l’utilisation ultime d’antimicrobiens alternatifs pour le traitement des infections CoNS, tels que le glycopeptide vancomycine. Entre-temps, des descriptions de sensibilité et de résistance réduites à la vancomycine ont été rapportées au cours des dernières décennies.,6 la sensibilité réduite à la vancomycine peut être liée à des modifications métaboliques telles que l’accélération de la synthèse du peptidoglycane, entraînant un épaississement de la paroi cellulaire. Dans ce processus, la vancomycine n’est pas capable d’inhiber la synthèse du peptidoglycane, car elle est épuisée en raison de la plus grande disponibilité des sites D-alanyl-D-alanine.,7
compte tenu de la recrudescence des infections hospitalières causées par les CoNS, cette étude visait à caractériser les souches résistantes à l’oxacilline et à déterminer la sensibilité aux antimicrobiens d’une collection de 20 ans d’isolats d’hémoculture CoNS provenant de patients hospitalisés à L’Hôpital das Clínicas de Botucatu.
méthodes et matériels
Trois Cents souches de CoNS ont été isolées dans des hémocultures de patients hospitalisés à L’Hôpital de Botucatu das Clínicas-Paulista State University (UNESP)., Les isolats ont été collectés de 1990 à 2009 et conservés dans le Laboratoire de collecte de Culture du Département de microbiologie et D’Immunologie de L’Institut des Biosciences de Botucatu – UNESP. Les critères de sélection ont considéré une prévalence moyenne des événements de 35%, avec une marge d’erreur de 5% et un intervalle de confiance de 95%.
Les souches ont été isolées selon Koneman et coll.8 isolats de gélose sanguine ont été soumis à une coloration de Gram pour l’observation de la morphologie de la colonie et le test de catalase a été effectué pour la confirmation du genre Staphylococcus., Des souches staphylococciques ont été soumises au test de coagulase pour la différenciation des groupes coagulase négative et coagulase positive. Les isolats à Coagulase négative ont été soumis à des tests biochimiques pour l’identification phénotypique des espèces. L’identification génotypique a été réalisée à l’aide d’amorces dessinées sur des séquences conservées adjacentes aux gènes 16S et 23S, par ITS-PCR (internal transcribed spacer–polymerase chain reaction), décrit par Couto et al.9 l’efficacité de L’Amplification a été surveillée par électrophorèse dans 3% d’agarose métaphorique et colorée avec SYBR Safe., The following lineages of international reference were used: S. auricularis ATCC 33753, S. capitis subsp. capitis ATCC 27843, S. capitis subsp. urealyticus ATCC 49325, S. caprae ATCC 35538, S. cohnii ATCC 49330, S. cohnii subsp. cohnii ATCC 29974, S. epidermidis ATCC 12228, S. epidermidis ATCC 35983, S. hemolyticus ATCC 29970, S. hominis ATCC 27844, S. hominis subsp. novobiosepticus ATCC 700237, S. lentus ATCC 700403, S. lugdunensis ATCC 700328, S. saprophyticus ATCC 15305, S. schleiferi subsp. schleiferi ATCC 43808, S. sciuri subsp. sciuri ATCC 29062, S. simulans ATCC 27851, S., xylosus ATCC 29979 et S. warneri ATCC 10209.
extraction D’ADN
Le kit Illustra (GE Healthcare) a été utilisé pour l’extraction D’ADN. Les étapes comprenaient une digestion initiale des cellules staphylococciques avec du lysozyme (10 mg/mL) et de la protéinase K (20 mg/mL). Ensuite, 500µL du tampon d’extraction ont été ajoutés au mélange, qui a été centrifugé à 10 000 x g pendant 4min. Le surnageant a été transféré dans une colonne et centrifugé à 5 000 g pendant 1 min. Le fluide a été rejeté et 500µl de tampon d’extraction ont été ajoutés à la colonne., Après la centrifugation et le rejet du fluide recueilli, 500µl de tampon de lavage ont été ajoutés à la colonne, qui a été soumise à la centrifugation à 20 000 g pendant 3min. Ensuite, la colonne a été transférée dans un tube de 1,5 mL et l’élution a été effectuée en utilisant 200µL D’eau MilliQ réchauffée à 70°C.
la détection du gène mecA
PCR a été réalisée pour la détection du gène mecA. Les réactions ont été effectuées en utilisant un protocole décrit par Murakami et al.10 l’efficacité de l’amplification a été contrôlée par électrophorèse dans un gel d’agarose à 2% teinté de SYBR Safe.,
détermination du Cemcc
le type de Cemcc a été déterminé sur des souches mecA-positives. Les réactions ont été réalisées en utilisant un protocole décrit par Oliveira et al.11 et modifié par Machado et coll.12
la PCR Multiplex a été réalisée dans 50µL de volume de réaction avec 1x tampon enzymatique, 1.,25 U Taq polymerase DNA, 200 μM dNTP Mix, and the following primers: 10pmol of RIF2 F2 (TTCGAGTTGCTGATGAAGAAGG) and CIF2 R2 (ATTTACCACAAGGACTACCAGC), 6pmol of KDP F1 (AATCATCTGCCATTGGTGATGC) and KDP R1 (CGAATGAAGTGAAAGAAAGTGG), 5pmol of DCS F2 (CATCCTATGATAGCTTGGTC) and DSC R1 (CTAAATCATAGCCATGACCG), 5pmol of RIF4 F3 (GTGATTGTTCGAGATATGTGG), and RIF4 R9 (CGCTTTATCTGTATCTATCGC). For each reaction, 10μL DNA was added. The cycle sequencing reactions were performed at 92°C for 3min, followed by 30 cycles of 92°C for 1min, 56°C for 1min, and 72°C for 1min and 30s., L’efficacité de l’amplification a été contrôlée par électrophorèse dans un gel d’agarose à 2% teinté de SYBR Safe.
détermination de la concentration inhibitrice minimale (CMI) par le test E
la sensibilité in vitro des souches de CoNS a été testée pour les antimicrobiens suivants: oxacilline, vancomycine, daptomycine, linézolide, quinupristine / dalfopristine et tigécycline. Le CIC de ces médicaments a été déterminé par le test E. Les critères utilisés pour la classification de sensibilité étaient les suivants: oxacilline
µg / mL (sensible) pour les inconvénients, sauf S., lugdunensis (sensible ≤2µg/mL et résistant ≥4µg/mL), vancomycine µg/mL (sensible), 8–16µg/mL (intermédiaire résistant) et >32µg/mL (résistant); linézolide ≤4µg/mL (sensible); daptomycine ≤1µg/mL; quinupristine/dalfopristine ≤1µg/mL (sensible), 2µg/ml (intermédiaire), et ≥4µg/ml (résistant); tigecycline ≤0.5 µg/ml.,13 test de dépistage pour la détection d’une sensibilité réduite à la vancomycine
afin de détecter une sensibilité réduite à la vancomycine, un test de dépistage sur gélose préparé avec de la gélose pour perfusion Brain Heart (BHI) et 4µg/mL, 6µg/mL, 8µg/mL et 16µg/mL de vancomycine a été utilisé. La souche de référence S. aureus ATCC 29213, sensible à la vancomycine, a été utilisée comme témoin négatif, et la souche E. faecalis ATCC 51299, résistante à la vancomycine, comme témoin positif. Taches d’un 2.,0 inoculum de McFarland ont été ajoutés à la plaque de gélose et incubés à 35°C pendant 24h, et la croissance d’au moins une colonie a été considérée comme un résultat positif.
analyse de l’épaisseur de la paroi cellulaire
Les souches CoNS présentant une sensibilité réduite à la vancomycine ont été soumises à la microscopie électronique de transmission pour analyse de l’épaisseur de la paroi cellulaire. Les souches ont été cultivées dans un bouillon BHI et incubées à 37° C pendant 24h.dans un microtube, 1000µl d’un bouillon de culture CoNS ont été centrifugées pendant une minute à 12 000 tr / min. Après avoir éliminé le surnageant, les souches ont été fixées dans une solution de Karnovsky (2.,5% de glutaraldéhyde dans un tampon de phosphate 0,1 M) pendant quatre heures. Les échantillons ont été retirés du fixateur et lavés trois fois pendant cinq minutes dans de l’eau distillée. Ensuite, les échantillons ont été immergés dans du tétroxyde d’osmium à 0,5% pendant 40 minutes, avant d’être lavés trois fois pendant 10 minutes dans de l’eau distillée. Les échantillons ont été déshydratés en utilisant des concentrations croissantes d’alcool: deux fois pendant 10min sur 7.,5% d’alcool; deux fois pour 10min à 15% d’alcool; deux fois pour 10min à 30% d’alcool; deux fois pendant 10 minutes dans 50% d’alcool; trois fois pendant 15min dans de l’alcool à 70%, deux fois pendant 15min à 90% d’alcool; deux fois pendant 10 minutes à 100% d’alcool. Après la déshydratation, les souches ont été montées et les échantillons métallisés. Les analyses ont été réalisées en microscopie électronique Tecnai Spirit Fei Company et les images ont été obtenues à un grossissement de 30000×.,
Résultatsidentification des isolats
Le tableau 1 présente la source des isolats D’espèces CoNS, ainsi que les données démographiques des patients atteints de bactériémie.
Détection du gène mecA et caractérisation du SCCmec détermination de la concentration minimale inhibitrice (CMI)
Les CMI des antimicrobiens utilisés pour le traitement des infections par CoNS ont été déterminés par le test E. La résistance à l’oxacilline a été trouvée dans 206 souches CoNS (68,7%) et 25 étaient résistantes à la tigécycline (8,3%). Une résistance à la tigécycline a été détectée chez 4,5% de S. epidermidis, 13,8% de S. hemolyticus, 13% de S., hominis, et 11,1% de S. lugdunensis. Pour la quinupristine / dalfopristine, un isolat de S. epidermidis présentait une résistance intermédiaire avec une CMI de 2µg/mL, et un S. epidermidis et un S. hemolyticus avec des CMI de 3µg/mL. La sensibilité et la spécificité de l’oxacilline MIC par le test E par rapport à la présence de mecA étaient respectivement de 87,4% et 82,3%.
en ce qui concerne les espèces CoNS, la distribution des paramètres MIC50 et MIC90 a révélé des taux élevés de résistance à l’oxacilline chez S. hemolyticus., C’était la seule espèce contre pour laquelle la concentration minimale pour inhiber 50% des isolats était >256µg/mL, reflétant le taux élevé de résistance de S. hemolyticus à l’oxacilline (Tableau 3).
La gamme de CMI de chaque antimicrobien a été comparée au type SCCmec. Les isolats typés comme SCCmecI présentaient des MICs de tigécycline plus élevés et ceux classés comme SCCmecII présentaient les MICs d’oxacilline les plus élevés. Les souches de SCCmecIV ont montré les valeurs de CMI les plus élevées pour la vancomycine, le linézolide, la daptomycine et la quinupristine/dalfopristine (Tableau 4).
Determination of the MIC range according to SCCmec.
| MIC range (μg/ml) | ||||
|---|---|---|---|---|
| Antimicrobial | SCCmec I | SCCmec II | SCCmec III | SCCmec IV |
| Oxacillin | 0.094≥256 | 6≥256 | 0.,125≥256 | 0.125≥256 |
| Vancomycin | 0.25–2 | 0.38–2 | 0.125–2 | 0.75–2 |
| Linezolid | 0.064–1 | 0.19–0.5 | 0.047–0.75 | 0.064–1 |
| Daptomycin | 0.016–025 | 0.016–0.094 | 0.019–0.5 | 0.047–0.75 |
| Quinupristin/Dalfopristin | 0.025–1.5 | 0.094–0.5 | 0.064–1 | 0.125–2 |
| Tigecycline | 0.016–2 | 0.023–0.75 | 0.016–0.75 | 0.,032-1 |
MIC, la Concentration Inhibitrice Minimale.
Une évaluation des CMI d’oxacilline et de vancomycine a été réalisée sur une période de 20 ans, divisée en trois périodes selon la distribution des gammes de CMI d’oxacilline et de vancomycine. Pour l’oxacilline, dans la première période (1990-2001), des valeurs plus élevées de CMI ont été observées, mais indiquant toujours une susceptibilité., Dans les autres périodes (2002-2006 et 2007-2009), une diminution des valeurs de CMI a été observée, indiquant toujours une susceptibilité, ainsi qu’une augmentation des taux de résistance. En ce qui concerne la vancomycine, la même évaluation a mis en évidence des différences significatives dans les trois périodes entre les souches ayant des CMI de 0,047 µg/mL à 0,38 µg/mL et de 0,5 µg/mL à 2,0 µg/mL (Fig. 1).
détermination de la sensibilité réduite à la vancomycine
Une croissance de 214 (71,3%) souches a été observée sur la plaque BHI avec 4µg/mL de vancomycine, 89 (29,7%) sur la plaque avec 6µg / mL de vancomycine et 8 (2.,6%) isolats sur la plaque de vancomycine de 8µg/mL. Aucun des isolats n’a poussé dans le milieu avec 16µg / mL de vancomycine. Il n’y avait pas de différences significatives dans les CMI entre les souches qui ne présentaient pas de croissance sur la plaque complétée par de la vancomycine et celles qui poussaient sur la plaque de vancomycine à 4µg/mL. Parmi ceux qui ont montré une croissance sur le milieu avec 6µg/mL de vancomycine, la gamme de CMI était de 0,5–2µg / mL. Parmi ceux qui ont augmenté sur 8µg/mL de vancomycine, la gamme de CMI était 0.75–24µg / mL. Les souches cultivées sur 8µg/mL de vancomycine étaient quatre S. epidermidis, une S. hemolyticus, deux S. hominis et une S., capitis, tous résistants à l’oxacilline, et S. epidermidis et S. capitis étant porteurs de SCCmec III, S. hominis de SCCmec I et S. hemolyticus avec SCCmec non typé.
analyse de l’épaisseur de la paroi cellulaire de souches présentant une sensibilité réduite à la vancomycine
l’épaisseur de la paroi cellulaire sous division cellulaire a été mesurée chez huit souches (les valeurs sont présentées en nanomètres, moyenne±écart-type). Les quatre souches de S. epidermidis ont présenté des valeurs moyennes de 21.66±1.4, 20.12±0.9, 24.24±1.3, et 14,20±1,3 nm. L’isolat de S. hemolyticus a montré une épaisseur de paroi cellulaire de 24,88±1,7 nm. Pour le S., isolat de capitis, l’épaisseur de la paroi cellulaire était de 19,33±1,8, et pour les deux isolats de S. hominis, les valeurs étaient de 17,68±2,1 et 24,92±1,5. À titre de comparaison, les souches de référence utilisées S. epidermidis ATCC 12228 et S. hemolyticus ATCC29970 ont montré des épaisseurs de paroi cellulaire de, respectivement, 14,61±1,2 et 14,73±0,7 nm. La comparaison des souches étudiées avec les témoins a montré des valeurs plus élevées, allant de 17 nm à 24,92 nm, à l’exception d’un isolat de S. epidermidis, qui présentait une valeur plus faible par rapport aux témoins (Fig. 2).,
contre l’évolution de la bactériémie
l’évolution possible de la bactériémie vers d’autres infections, telles que septicémie, pneumonie, infection des voies urinaires, méningite, péritonite, entérocolite nécrosante, omphalite et abcès cérébral, a été suivie chez 52 patients admis dans les services de néonatalogie. Parmi ceux-ci, chez 25 patients (46,3%), le même CoNS isolé à partir d’une hémoculture a été confirmé comme étant l’agent étiologique de l’infection. Dans six (11,1%), le CoNS était l’agent possible de l’infection, et dans 23 (42,6%) le CoNS n’était pas lié à l’infection du patient (fig. 3).,
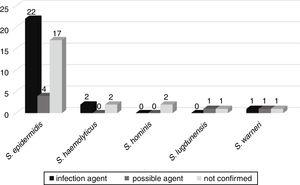
corrélation des CoNS isolés des hémocultures en tant qu’agents étiologiques possibles des infections causées chez les patients néonatals.
Le gène mecA a été détecté dans 33 (61.1%) de ces Inconvénients, avec 20 (60,6%) des Inconvénients associés aux infections plus graves ou les agents possibles de ces infections.
Discussion
les inconvénients sont considérés comme l’une des principales causes de bactériémie., L’importance de ces bactéries a augmenté dans l’environnement hospitalier au cours des dernières années, principalement en raison de la résistance aux antimicrobiens. Dans le présent travail, 300 souches de CoNS isolées à partir d’hémocultures de patients hospitalisés à L’Hôpital des cliniques de Botucatu, sur une période de 20 ans, ont été étudiées. Ces isolats ont été caractérisés en ce qui concerne leur sensibilité aux antimicrobiens.
la détection mecA a déterminé la résistance à l’oxacilline dans 78,5% des CoNS, et la valeur MIC50 (2µg/mL) a été indicative de la résistance à ce médicament, confirmant la faible sensibilité de ces bactéries aux agents β–lactamines., Des travaux antérieurs ont démontré que depuis les années 1970, les isolats de CoNS présentent des taux de résistance à l’oxacilline plus élevés que ceux de S. aureus.14,15 il a été démontré que les taux de résistance aux β–lactamines variaient de 65% à 95% dans les hôpitaux du Brésil et d’autres pays.2,16,17 les taux les plus élevés de résistance à l’oxacilline ont été observés chez S. epidermidis, suivi de S. hemolyticus, S. hominis et S. capitis. Des résultats similaires ont été décrits dans des études antérieures, qui ont montré une résistance à L’oxacilline chez 97% des S. epidermidis entre 1999 et 2001 dans une unité de soins intensifs néonatals (USIN),18 chez 96% des S., hemolyticus isolé au Brésil, 19 et dans 100% de S. hominis isolé dans une USIN D’Espagne.20 en ce qui concerne S. capitis, des résultats divergents ont été trouvés dans les études menées par Caierão et al.21
bien qu’à une fréquence plus faible, le gène mecA a été détecté chez S. warneri et S. lugdunensis. S. warneri a été décrit avec des taux de 33,3% de résistance à l’oxacilline chez NICUs,22 et la première description de mecA chez S. lugdunensis a été dans l’étude menée par Kawaguchi et al.23 malgré les faibles taux de résistance, 24 détermination de la sensibilité aux antimicrobiens de S., lugdunensis est important, non seulement en raison de ses implications cliniques, puisque cette espèce est la plus agressive des inconvénients, mais aussi pour la mise en place d’un traitement précoce avec des antimicrobiens adéquats et de bons résultats cliniques.25
La caractérisation du SCCmec a montré la présence de types I-IV dans les isolats, le SCCmecIII étant le plus fréquemment détecté dans les souches étudiées, principalement chez S. epidermidis, la seule espèce porteuse du type IV., SCCmec de type III est le plus grand de tous, codifie pour plusieurs gènes associés à la résistance et est le contre le plus souvent isolé à partir de spécimens hospitaliers. S. epidermidis est le principal colonisateur de la peau humaine et le plus souvent détecté dans les sites d’infection. La pression sélective dans l’environnement hospitalier entraîne la dissémination de souches SCCmec III, qui sont associées à des infections graves.26 SCCmec de type IV a été associé à Staphylococcus spp., et a été décrit pour la première fois dans une souche de S. epidermidis, un fait qui implique son transfert de S., epidermidis à S. aureus. Un transfert à moindre coût de SCCmec IV en raison de sa petite taille entraînerait probablement une incidence plus élevée d’infections causées par les porteurs de SCCmec IV.12,27 SCCmecII a présenté une association avec S. hemolyticus, semblable aux conclusions de Machado et al., 12 dont les travaux n’ont détecté que le type II chez cette espèce.
l’alternative pour le traitement des staphylocoques résistants à l’oxacilline est la vancomycine, un glycopeptide utilisé pour la première fois en 1958 dans les infections invasives. Il existe cependant des descriptions de résistance et de sensibilité réduite à ce médicament6,bien qu’instable.,28 les résultats des travaux en cours ont montré que, à l’exception de l’oxacilline, la vancomycine était le médicament avec les valeurs les plus élevées de MIC50 et MIC90. Malgré la sensibilité totale de la collection, les valeurs de CMI ont été les plus élevées au cours de la période de 20 ans, et une sensibilité réduite a été détectée chez certaines souches de S. epidermidis, S. hemolyticus, S. hominis et S. capitis. Dans les études réalisées par Natoli et coll., 29 une sensibilité réduite à la vancomycine a été détectée avec une fréquence de 5,4% parmi les isolats de CoNS, chez les espèces de S. epidermidis et de S. hemolyticus., Selon les mêmes auteurs, les colonies de staphylocoques cultivées sur gélose à la vancomycine peuvent entraîner une hétérorésistance des glycopeptides, qui peut être un précurseur de la résistance aux glycopeptides, provoquant des infections compliquées et un échec du traitement. Cela devrait être pris en considération dans les décisions thérapeutiques.30
l’augmentation de la proportion de CoNS résistants à l’oxacilline et la diminution de leur sensibilité à la vancomycine soulignent l’importance des études impliquant d’autres choix thérapeutiques., La tigécycline, une glycylcycline semi-synthétique à large spectre Gram positif et négatif, est considérée comme un médicament ayant une excellente activité contre l’oxacilline sensible et résistante contre 14,29 malgré nos données montrant des taux de 8,3% de résistance,le plus souvent chez S. hemolyticus, suivi de S. hominis, S. lugdunensis et S. epidermidis. Des résultats MIC90 similaires ont été décrits pour la tigécycline, sans différence entre les contre résistants à l’oxacilline et sensibles.31 dans les études menées par Natoli et al.29 la tigécycline a démontré une bonne activité contre contre., Selon ces auteurs, la tigécycline n’est pas recommandée pour le traitement de la bactériémie et son utilisation doit être limitée afin de préserver son activité contre les bactéries Gram négatives multi-résistantes. Les Mutations du gène ribosomique S10 et des gènes rpsJ et mepA semblent être associées à la résistance à la tigécycline.32,33
La quinupristine / dalfopristine est une streptogramine appartenant au groupe macrolide-lincosamide-streptogramine., La combinaison de quinupristine et de dalfopristine est synergique et généralement bactéricide lorsque ces agents sont comparés individuellement ou comparés à des antimicrobiens macrolides similaires.34 dans ce travail, bien que présentant une excellente efficacité dans la majorité des contre étudiés, une résistance intermédiaire à ce médicament a été détectée chez deux S. epidermidis et un S. hemolyticus. Dans l’étude menée par Mendes et coll., 35 quinupristine / dalfopristine ont démontré une excellente efficacité pour les contre, les souches étaient entièrement sensibles, avec des valeurs MIC90 de 0,38 µg / mL pour les souches sensibles à l’oxacilline et 0.,75µg / mL pour les souches résistantes à l’oxacilline. En outre, Venkatesh et al.36 décrit une bonne efficacité de la quinupristine / dalfopristine pour les souches sensibles et résistantes à l’oxacilline.
la daptomycine, un antimicrobien étudié depuis des décennies, a été remise en service en 2006, pour le traitement de la bactériémie et de l’endocardite causées par le staphylocoque.37 dans la présente étude, la daptomycine a montré une excellente efficacité pour les isolats CoNS, avec de faibles valeurs MIC50. Olivares et coll.37 a également vérifié la sensibilité de tous les CoNS à la daptomycine, avec une faible variation des valeurs de CMI., Dans un travail étudiant 1126 contre, dirigé par Critchley et al., 38 la daptomycine était active sur une plage de CMI comprise entre 0,015 et 2,0 µg/mL, et la CMI 90 était de 0,5 µg/mL. Selon ces auteurs, les agents les plus actifs dans ces analyses étaient la daptomycine et la quinupristine/dalfopristine, ce qui souligne la pertinence de ces antimicrobiens dans le traitement des infections causées par des CoNS.
le linézolide, une oxazolidinone synthétique potentiellement active contre plusieurs bactéries, est un autre médicament qui a démontré une bonne efficacité contre les CoNS.,39 le linézolide est devenu important pour le traitement thérapeutique des infections chroniques par contre, malgré la description de la résistance. Au Brésil, le premier cas de résistance aux linézolides a été décrit en 2006, sur un isolat clinique de MRSA40 et, parmi les inconvénients, chez S. epidermidis, S. hominis et S. hemolyticus.41 dans les travaux menés par Olivares et al., 37 malgré la détection de sept souches résistantes aux linézolides, ce médicament a présenté des valeurs de CMI efficaces dans les souches CoNS. D’autre part, une épidémie de linézolide résistant S., des clones d’epidermidis contenant des mutations dans les protéines ribosomiques L3 et L4, ainsi que le plasmide cfr, récemment identifié en France, ont été rapportés.42
S. hemolyticus était l’espèce qui présentait les valeurs de CMI d’oxacilline les plus élevées, et des concentrations supérieures à 256µg / mL étaient nécessaires pour inhiber 50% des isolats. En plus de l’oxacilline, cette espèce présentait des valeurs de CMI plus élevées pour la tigécycline et la plupart des antimicrobiens testés. Plusieurs travaux montrent des taux élevés de résistance aux antimicrobiens utilisés pour le traitement des infections causées par S., hémolytique, en particulier la méthicilline et les glycopeptides. Certains auteurs décrivent une forte prévalence de gènes codant pour la Résistance Aux β-lactamines et aux aminoglycosides, ainsi qu’une proportion significative d’isolats avec des valeurs de CMI proches du point de rupture de la resistance,43,44 soulignant l’importance de S. hemolyticus en tant qu’agent pathogène multirésistant.
l’association de la gamme de CMI avec les types SCCmec a démontré des CMI plus élevés pour la vancomycine, le linézolide, la daptomycine et la quinupristine/dalfopristine chez S. epidermidis porteur de SCCmecIV., Les gènes liés à la résistance à ces antimicrobiens sont médiés par des plasmides, trouvés principalement dans des souches résistantes à la méthicilline d’origine hospitalière.45 SCCmec IV est caractéristique des isolats communautaires, en particulier en raison de sa taille et de son faible coût adaptatif. Des études suggèrent que l’acquisition de gènes de résistance a une origine non clinique environnementale, étant donné la grande diversité des porteurs de gènes de résistance dans l’environnement naturel.46,47 en outre, SCCmecIV n’a été détecté que chez S. epidermidis, qui, en tant qu’espèce la plus commune dans la peau humaine, est la plus influencée par la pression sélective., Les MICs plus élevés par rapport à d’autres antimicrobiens peuvent également être liés à la pression sélective, car le niveau d’exposition à ces médicaments dans l’environnement hospitalier biaiserait la sélection de souches à sensibilité réduite et résistantes, car plusieurs événements de mutation, recombinaison génétique et modifications de la physiologie microbienne sont nécessaires pour générer des changements phénotypiques.,48
des modifications métaboliques et physiologiques liées à la pression sélective ont pu être observées chez des souches cultivées sur gélose de vancomycine à 8µg / mL, car l’épaississement de la paroi cellulaire a été constaté dans la plupart des isolats de CoNS en raison de l’augmentation de la synthèse du peptidoglycane. Ce mécanisme est plus avantageux par contre que l’acquisition de l’opéron van, qui est un médiateur de la résistance à la vancomycine. L’opéron van est acquis par transfert horizontal de gènes et son expression n’est stimulée qu’en présence de glycopeptide., Ce stimulus entraîne un coût adaptatif très élevé en présence de vancomycine, étant désavantageux pour les inconvénients dans un milieu de vancomycine.48,49 en ce qui concerne l’acquisition d’autres gènes de résistance en CoNS, tels que le gène mecA, le coût adaptatif initial est adouci par des mutations supplémentaires compensatoires pour les « coûts”de résistance. Récemment, des mutations ponctuelles dans des gènes tels que vraR ont été associées à une sensibilité réduite à la vancomycine et à un épaississement de la paroi cellulaire chez les staphylocoques.,50
La présente étude a montré un taux élevé de bactériémie causée par des CoNS dans les unités néonatales et pédiatriques, impliquant des enfants de moins d’un an, les principaux agents étant S. epidermidis, S. hemolyticus, S. hominis, S. lugdunensis et S. warneri. S. capitis n’ont pas été isolés dans ces unités, étant des agents de bactériémie chez les adultes admis au service de médecine interne, à la gastro-entérologie, à la neurologie et aux urgences.,
comme l’étude impliquait une bactériémie causée par des staphylocoques isolés sur plus de 30 ans, une grande partie des données associées aux dossiers médicaux des patients n’a pas pu être récupérée, il n’a donc pas été possible d’associer le résultat de l’infection de tous les patients, ce qui est une limitation de l’étude actuelle. Les données obtenues auprès de patients dans des unités néonatales ont indiqué l’importance clinique de la bactériémie CoNS, qui peut évoluer vers des maladies nécessitant des soins spécialisés.
La connaissance de la résistance aux antimicrobiens est d’une grande importance pour le traitement correct des infections causées par les CoNS., De plus, les facteurs de virulence de ces bactéries et l’immunité des patients sont des facteurs qui contribuent à la capacité du micro-organisme à provoquer des infections plus graves.
les résultats de notre étude sur 20 ans ont montré une fréquence élevée de résistance aux antimicrobiens chez les CoNS, ce qui reflète l’utilisation excessive de ces médicaments., Outre les modifications métaboliques et physiologiques qui pourraient conduire à une sensibilité réduite aux antimicrobiens, leur condition de bactéries commensales humaines en font des transporteurs idéaux et un réservoir efficace de gènes de résistance, en particulier les éléments à faible coût, tels que SCCmec.45 les antimicrobiens qui ont donné les meilleurs résultats devraient être utilisés de manière à préserver leur efficacité et à prévenir la résistance.,
conflits d’intérêts
Les auteurs déclarent que la recherche a été menée en l’absence de toute relation commerciale ou financière pouvant être interprétée comme un conflit d’intérêts potentiel.