Einführung
Im Gegensatz zu Tieren, die sich bewegen können, um die widrige Umgebung zu vermeiden, weisen die Sitzpflanzen eine hochentwickelte Anpassung an die komplizierten Umweltbedingungen auf. Um diese tiefgreifende Anpassungsfähigkeit zu erreichen, ist die Kommunikation zwischen den Zellen notwendig. Die Kommunikation von Zelle zu Zelle in Pflanzen umfasst eine robuste intrazelluläre Signalverarbeitung und komplizierte interzelluläre Signalnetze., Bis jetzt wurden mindestens neun Signalstoffe namens Pflanzenhormone entdeckt, darunter Auxin, Brassinosteroid (BR), Cytokinin, Gibberelline (GA), Ethylen, Jasmonsäure (JA), Strigolacton (SL), Abscisinsäure (ABA) und Salicylsäure (SA) (Druege et al., 2016; Verma et al., 2016). Die genetischen und physiologischen Studien haben die kritischen Rollen und funktionellen Mechanismen dieser oben genannten Hormone im Pflanzenwachstum und in der Entwicklung aufgedeckt (Gray, 2004)., Basierend auf den vorherigen Studien funktionieren Auxin, BR, GA, SL und Cytokinin hauptsächlich während des normalen Pflanzenwachstums und der normalen Pflanzenentwicklung, während ABA, Ethylen, JA und SA eine wichtige Rolle bei der Reaktion des Pflanzenwachstums auf verschiedene biotische und abiotische Belastungen spielen (Pieterse et al., 2009; Santner et al., 2009; Denance et al., 2013). Und auch einige dieser Hormone haben eine doppelte Rolle, zum Beispiel spielt ABA auch eine wichtige Rolle bei der Samenentwicklung und-ruhe (Seo und Koshiba, 2002)., Obwohl jedes Hormon in bestimmten Aspekten eine vorherrschende Rolle spielt, haben sich viele Hormone überlappende Aktivitäten, und die Wechselwirkungen verschiedener Hormone steuern viele Entwicklungsaspekte und das Wachstum als Reaktion auf endogene entwicklungs-und exogene Signale.
Auxin und BR sind zwei Hauptklassen von wachstumsfördernden Hormonen., BR, eine Gruppe pflanzenspezifischer Steroidhormone, die mit anderen Phytohormonen wie Auxin, Cytokinin, Ethylen, GA, JA und SA interagieren und eine breite Palette von Pflanzenwachstums-und Entwicklungsprozessen regulieren können, einschließlich Samenkeimung, Zelldehnung, Gefäßdifferenzierung, Stomatenbildung und-bewegung, Blüte und männliche Fruchtbarkeit (Saini et al., 2015). Interessanterweise wird jeder dieser Prozesse auch von Auxin gesteuert, was darauf hindeutet, dass diese beiden Hormone zusammenspielen, um die Pflanzenentwicklung zu kontrollieren., In dieser Übersicht werden wir die Signaltransduktion von Auxin und BR auf der Grundlage der jüngsten Fortschritte skizzieren und den Übersprechen zwischen Auxin und BR vermitteltem Pflanzenwachstum und-entwicklung überprüfen.
Auxin Signalweg
Auxin wurde zuerst als Pflanzenhormon wegen seiner Rolle im Pflanzentropismus gegen Schwerkraft oder Lichtreize erkannt., Später wurde Auxin chemisch als Indol-3-Essigsäure identifiziert und spielte nachweislich eine wesentliche Rolle bei einer Vielzahl von Pflanzenentwicklungs-und physiologischen Prozessen, einschließlich Embryogenese, Organogenese, Gefäßdifferenzierung, Wurzel-und Triebentwicklung, tropisches Wachstum und Fruchtentwicklung (Estelle, 2011).
Unter Verwendung der genetischen Analyse in Arabidopsis wurde der molekulare Mechanismus, der der Auxin-Signaltransduktion zugrunde liegt, gut untersucht. TRANSPORT INHIBITOR RESPONSE1 (TIR1) war der erste identifizierte nukleare Rezeptor von Auxin (Ruegger et al., 1998; Dharmasiri et al., 2005)., TIR1 kodiert ein Kernprotein, das zum F-Box-Protein als Untereinheit des SCF E3-Ubiquitinligase-Proteinkomplexes gehört (Gray et al., 1999, 2002; Hellmann et al., 2003; Quint et al., 2005) gibt es neben TIR1 drei weitere F-Box-Proteine, nämlich AUXIN-SIGNALISIERENDE F-BOX-Proteine (AFBs), die Auxin-bindende Aktivität zeigen und Auxin-Signalisierung in Arabidopsis vermitteln (Badescu und Napier, 2006). Der TIR1-Rezeptor kann mit einer Gruppe von AUX/IAA-Proteinen (Auxin/Indol-3-Essigsäure) interagieren (Dharmasiri et al., 2003)., AUX / IAA-Proteine sind negative Regulatoren der Auxin-Signalgebung und es gibt 29 Mitglieder von AUX/IAA, die im Arabidopsis-Genom kodiert sind. AUX / IAA-Proteine könnten mit der Klasse der Transkriptionsregulatoren, Auxin Response Factors (ARF), interagieren, um Transkriptionsreaktionen auf Auxin zu vermitteln. Unter hohem Auxin-Spiegel interagieren AUX/IAA-Proteine mit TIR1 als Corezeptor von Auxin und können durch den SCFtir1-Komplex ubiquitiniert und somit durch den Ubiquitin–Proteasom-Weg abgebaut werden (Gray et al., 2001; Lanza et al., 2012)., Nach der Zerstörung von AUX / IAA-Repressoren können die Auxin-Transkriptionsregulatoren ARFs, zu denen 23 Mitgliedschaften gehören, von der AUX/IAA-Repression befreit werden und somit die Auxin-Reaktionen durch Aktivierung oder Repression von Zielgenen vermitteln (Guilfoyle und Hagen, 2007). Die verschiedenen Sätze von F-Box-Protein und AUX / IAA oder ARFs schließen die Komplexität während der Auxin-Signaltransduktion ab (Goh et al., 2012; Guilfoyle, 2015; Salehin et al., 2015).,
Die koordinierte Wirkung von Aux / IAA-Transkriptions-Repressoren und ARF-Transkriptionsfaktoren erzeugt komplexe genregulatorische Netzwerke, über die auch in Physcomitrella berichtet wurde (Lavy et al., 2016). Kürzlich wurde festgestellt, dass CULLIN1 (CUL1) – Untereinheit der SCF mit TIR1 interagiert und somit TIR1-Substratstabilität und Auxin-Signalgebung reguliert (Wang et al., 2016). Die Wechselwirkung zwischen TIR1 und Aux/IAA wird auch durch die räumliche Konformation von Aux/IaaS beeinflusst, die durch eine Cyclophilin-Isomerase LRT2 in Reis gesteuert wird (Jing et al., 2015)., HITZESCHOCKFAKTOR 90 (HSP90) und das Co-Chaperon SGT1 interagieren jeweils mit TIR1 und regulieren somit die TIR1-Stabilität, was die Wechselwirkungen zwischen TIR1 und Aux/IAA und Auxin-Signalisierung beeinflusst (Wang et al., 2016).
Neben dem TIR1-abhängigen kanonischen Auxin-Signalweg wurde kürzlich berichtet, dass Auxin durch einen nicht-kanonischen Auxin-Signalmechanismus eine Vielzahl von Entwicklungsreaktionen hervorruft., In diesem nicht-kanonischen Auxin-Sensorprozess steuert ARF3/ETTIN die Genexpression durch Wechselwirkungen mit prozessspezifischen Transkriptionsfaktoren, die die Auxin-vermittelte Pflanzenentwicklungsvielfalt stark bereichern (Simonini et al., 2016, 2017).
BR Signalweg
BRASSINOSTEROID wurde zuerst in Pollen für seine Fähigkeit entdeckt, die Zelldehnung zu fördern. Später wurde festgestellt, dass BR eine Rolle in einer Vielzahl von Pflanzenwachstumsaspekten spielt und auf biotische und abiotische Belastungen reagieren kann., Heutzutage wurde der Signaltransduktionsweg weitgehend durch Kombinationen verschiedener Methoden geklärt, einschließlich Molekulargenetik, Biochemie, Proteomik und Genomik usw. Die Zell-Oberflächen-Kinase BRASSINOSTEROID INSENSITIVE1 (BRI1) wurde als der Rezeptor von BR identifiziert, der an die extrazelluläre Domäne von BRI1 binden und seine Kinase-Aktivität aktivieren und somit eine Signalkaskade zur Regulierung der Transkription einschalten kann (Li und Chory, 1997; Wang et al., 2001; Kinoshita et al., 2005; Kim und Wang, 2010; Clouse, 2011; Hothorn et al., 2011; She et al., 2011; Oh et al., 2012)., Bei der Wahrnehmung von BR interagiert BRI1 mit der Co-Rezeptor-BRI1-ASSOZIIERTEN KINASE 1 (BAK1) und ihren homolog SOMATISCHEN EMBRYOGENESISREZEPTORKINASEN (SERKs), um einen aktiveren BR-Rezeptorkomplex zu bilden (Li et al., 2002; Nam und Li, 2002; Wang et al., 2005; Tang et al., 2008; Gou et al., 2012). Aktivierte BRI1-Phosphorylate zwei Substrate von Plasmamembran-verankerten rezeptorähnlichen zytoplasmatischen Kinasen: BRASSINOSTEROID-SIGNALKINASE1 (BSK1) und KONSTITUTIVES DIFFERENTIELLES WACHSTUM1 (CDG1) (Tang et al., 2008; Kim et al.,, 2011), die wiederum eine PP1-Phosphatase namens BRI1-SUPPRESSOR1 (BSU1) phosphoryliert, um BSU1 zu aktivieren, was zur BSU1-Dephosphorylierung und Inaktivierung der GSK3-ähnlichen Kinase BRASSINOSTEROID INSENSITIVE2 (BIN2) führt. Die Kinaseaktivität von BIN2 wird auch durch HISTON-DEACETYLASE HDA6 gehemmt, die an der K189 von BIN2 interagiert und deacetyliert., Wenn der BR-Spiegel niedrig ist, ist BRI1 aufgrund seines negativen Reglers, des BRI1-KINASE-INHIBITORS 1 (BKI1) und der Proteinphosphatase 2A (PP2A), ruhiggestellt, während BIN2-Phosphor zwei homologe BR-Transkriptionsfaktoren, BRASSINAZOL-RESISTANT1 (BZR1) und BZR2 (auch BES1 für BRI1-EMS-SUPPRESSOR 1 genannt), bindet (He et al., 2002; Wang et al., 2002; Yin et al., 2002; Mora-Garcia et al., 2004; Kim et al., 2009, 2011; Kim und Wang, 2010). Wenn BR-Spiegel hoch sind, wird BIN2 inaktiviert, und BZR1 und BZR2 werden durch PP2A dephosphoryliert und bewegen sich in den Kern, um die Expression von Tausenden von BR-Antwortgenen zu verändern (He et al.,, 2005; Yin et al., 2005; Sun et al., 2010; Tang et al., 2011; Yu et al., 2011).
Die Synergie zwischen BR-und Auxin-Signalisierung
Auxin-und BR-Signalwege spielen unterschiedliche Rollen, sie zeigten jedoch auch synergistische und voneinander abhängige Wechselwirkungen in einer Vielzahl von Entwicklungsprozessen. Zum Beispiel können sowohl Auxin – als auch BR-Signale die Zellexpansion fördern und synergistisch interagieren, um die Hypocotyldehnung zu fördern (Nemhauser et al., 2004)., Die Reaktion eines der beiden Pfade bei der Förderung der Hypokotyldehnung erfordert die Funktion des anderen und die Interdependenz zwischen BR – und Auxin-Pfaden (Nemhauser et al., 2004). Auxin erhöhte die Hypocotyllänge in Wildpflanzen, jedoch nicht in der BR-unempfindlichen Mutante bri1-116, und dieser Auxin-unempfindliche Phänotyp von bri1-116 wurde durch die dominante Funktionsverstärkungsmutante bzr1-1D unterdrückt, was darauf hinweist, dass BR oder aktives BZR1 für die Auxin-Förderung der Hypocotyldehnung erforderlich ist., Es wurde gefunden, dass die BR-Signalisierung mit DEM SUPPRESSOR VON PHYTOCHROM B4-3 (SOB3) konvergiert, um die Zelldehnung und das Hypocotylwachstum durch die Regulation der Auxin-induzierten KLEINEN AUXIN-UP-RNA19 (SAUR19) – Expression zu kontrollieren (Favero et al., 2017). Auf der anderen Seite wurde kürzlich festgestellt, dass der Auxin-regulierte Transkriptionsfaktor SMALL ORGAN SIZE 1 (SMOS1) die Zellexpansion durch die direkte Wechselwirkung mit SMOS2/DLT steuert, einem Mitglied der GRAS-Familie transkriptioneller Co-Regulatoren, das eine positive Rolle bei der BR-Signalisierung in Reis spielt (Kim et al., 2009; Tong et al., 2012; Hirano et al., 2017)., Auxin-verwandte Mutanten wie iaa3 und arf6 / arf8 waren weniger empfindlich gegenüber BR als Wildtyp für die Hypocotyldehnung und beseitigten die Überempfindlichkeit von bzr1-1D gegenüber Auxin, was darauf hindeutet, dass die BR-und BZR1-Förderung der Hypocotyldehnung ARF6/8 erfordert. Die genomweite ChIP-Seq-Analyse ergab, dass ARF6 durch CHIP-Seq-Analysen eine große Anzahl genomischer Ziele (etwa 50%) mit BZR1 und dem licht – /temperaturregulierten Transkriptionsfaktor PIF4 teilt (Oh et al., 2014)., BZR1 und PIF4 interagieren mit ARF6 und aktivieren gemeinsam genutzte Zielgene, indem sie während der Hypokotylen-Dehnung kooperativ an gemeinsam genutzte Zielgene binden (Oh et al., 2014) und viele dieser überlappenden Zielgene kodieren für Zellwandproteine, die an der Zellexpansion beteiligt sind.
Brassinosteroid und Auxin spielen auch eine wichtige Rolle bei der Aufrechterhaltung des apikalen Wurzelmeristems (RAM) (Durbak et al., 2012). Der WIDDER besteht aus einer kleinen Gruppe selten teilender Zellen, die als Ruhezentrum (QC) bekannt sind und von Stammzellen umgeben sind, aus denen die verschiedenen Toot-Gewebetypen hervorgehen., Die Aufrechterhaltung der Wurzelstammzellpopulation wird durch WUSCHEL-BEZOGENE HOMÖOBOX 5 (WOX5) reguliert (Sarkar et al., 2007). WOX5 ist durch Auxin-Signalisierung auf die QC beschränkt und erleichtert die ordnungsgemäße Expression der PLT-Gene (Aida et al., 2004; Ding, und Friml, 2010). Mutationen im BR-Rezeptorgen BRASSINOSTEROID INSENSITIVE 1 (BRI1) führen zu einem aberranten Zellzyklus Progression im RAM und verursachen eine kleinere RAMs (Gonzalez-Garcia et al., 2011; Hacham et al., 2011). Es ist bekannt, dass Auxin die Biosynthese von BR stimuliert (Chung et al.,, 2011), aber die Aktivität von BR beeinflusst nicht die Expression von PIN-Genen (Hacham et al., 2011). Die Wurzelspitzenphänotypen von BR-Mutanten zeigen nicht dasselbe wie die Auxin-Mutanten (Gonzalez-Garcia et al., 2011), was darauf hinweist, dass BR unabhängig von Auxin auf den RAM einwirken.
Brassinosteroid-und Auxin-Signale sind auch synergistisch bei der radialen Musterbildung von Gefäßbündeln erforderlich (Ibanes et al., 2009)., Durch die Kombinationen von mathematischer Modellierung und biologischen Experimenten ist Auxin Maxima, das durch asymmetrischen Auxin-Polartransport, aber nicht durch Veränderungen der Auxin-Spiegel, hergestellt wird, wichtig für die Positionierung der Gefäßbündel. Es wurde gezeigt, dass das BR-Signal als Fördersignal für die Anzahl der Zellen im provaskulären Ring dient, die mit Auxin-Maxima übereinstimmen. Somit steht die Etablierung einer periodischen Anordnung von Gefäßbündeln im Trieb unter der koordinierten Wirkung dieser beiden Pflanzenhormone (Ibanes et al., 2009)., Beide Signale sind auch an der Pflanzenwurzelentwicklung beteiligt und die Wechselwirkung von BR und Auxin wird durch BREVIS RADIX (BRX) während dieses Prozesses vermittelt. BRX ist wichtig für die ratenbegrenzende Biosynthese von BR und BR exogene Anwendung kann brx mutierte Defekte retten. Darüber hinaus ist die Auxin-responsive Genexpression bei Brx-Mutanten global beeinträchtigt, und die Expression von BRX wird stark durch Auxin induziert und durch BR unterdrückt, was bedeutet, dass BR-Biosynthese und Auxin-Signalisierung während der Wurzelentwicklung über eine Rückkopplungsschleife mit BRX verbunden sind (Mouchel et al., 2006).,
Brassinosteroide und Auxin spielen auch bei der lateralen Wurzelentwicklung eine synergistische Rolle. BRs funktionieren hauptsächlich bei der Initiation der lateralen Wurzel primordia, während Auxin sowohl für die Initiations-als auch für die Emergenzphase der lateralen Wurzelbildung erforderlich ist (Casimiro et al., 2001; Bhalerao et al., 2002; Benkova et al., 2003; Bao et al., 2004). Während dieser Prozesse erhöhen BRs die LRP-Initiation, indem sie den akropetalen Auxin-Transport in der Wurzel fördern, jedoch nicht den endogenen IAA-Spiegel beeinflussen (Bao et al., 2004)., Alle diese Berichte deuten darauf hin, dass der Übersprechen zwischen BR und Auxin spielt eine wichtige Rolle bei der Regulierung Pflanzenwachstum und-entwicklung.
BR reguliert die Auxin-Signalisierung
Neben der Interdependenz und Zusammenarbeit von Auxin-und BR-Signalen während der Pflanzenentwicklung könnte BR den Auxin-Signalweg auf mehreren Ebenen vermitteln. BZR1 interagiert mit ARF-Proteinen, um direkt auf mehrere Auxin-Signalkomponenten und Gene abzuzielen, die am Auxin-Stoffwechsel beteiligt sind, wie Transport und Signalisierung, einschließlich AUX/IAA, PINs, TIR1 und ARFs usw. (Sun et al., 2010)., Es wurde festgestellt, dass die Aux/IAA-Proteine BR Antworten und iaa7/axr2-1 und iaa17/axr3-3-Mutanten zeigten aberrante BR Empfindlichkeit und abweichende BR-induzierte Genexpression in einem organ-abhängigen Weise (Nakamura et al., 2006). Die Behandlung mit exogenem Brassinolid (BL) könnte die Expression von Auxin-responsiven Genen wie IAA5, IAA19, IAA17 usw. induzieren., und die Expression der oben genannten Gene wird in der BR-biosynthetischen Mutante de-etiolated2 (det2) herunterreguliert, was darauf hinweist, dass die funktionelle BR-Biosynthese teilweise für die Auxin-abhängige Genexpression erforderlich ist (Nakamura et al.,, 2003; Kim et al., 2006). Darüber hinaus beeinflusst BR auch den Auxin-Fluss, indem es die Expression von Auxin-Exportern wie PIN4 und PIN7 reguliert (Nakamura et al., 2004). Während Pflanzengravitropismusreaktionen könnte BRs die polare Akkumulation des Auxins und PIN2 in der Wurzelmeristemzone verstärken und somit die Umverteilung von Auxin von der Wurzelspitze zu den Dehnungszonen beeinflussen und zu der Differenz der IAA-Spiegel sowohl auf der oberen als auch auf der unteren Seite der Wurzeln führen, um Pflanzengravitropismus zu induzieren., Während dieses Prozesses spielt BR-ROP2 eine wichtige Rolle bei der Modulation der funktionellen Lokalisierung von PIN2 durch die Regulierung der Montage/Wiedermontage von F-Aktinen (Li et al., 2005). Weitere Studien zeigten, dass eine verminderte BL-Wahrnehmung und / oder-konzentration CYP79B2 induzieren könnte, das Gen, das für ein Enzym kodiert, das Tryptophan in Indol-3-Acetaldoxim umwandelt und somit die Verteilung beeinflusst (Kim et al., 2007).
Darüber hinaus wurde festgestellt, dass das BR-Signal die Auxin-Signalausgabe durch seinen negativen Regler GSK3 Kinase BIN2 regulieren kann., Der Auxin-Reaktionsfaktor ARF2 wurde als BIN2-interagierendes Protein in einem Hefe-Zwei-Hybrid-Bildschirm identifiziert, und der Kinase-Assay zeigte, dass BIN2 ARF2 phosphorylieren konnte. Die Phosphorylierung von ARF2 führt zum Verlust seiner DNA-Bindungsfähigkeit und Repressionsaktivität der Zielgene (Vert et al., 2008). ARF2 ist ein BZR1-Zielgen und seine Expression wird durch BR-Behandlung reduziert (Sun et al., 2010)., Zusätzlich kann BIN2 ARF7 und ARF19 phosphorylieren, um ihre Wechselwirkung mit AUX/IaaS zu unterdrücken und dadurch die Transkriptionsaktivität an ihren Zielgenen LATERALE ORGANGRENZEN zu verbessern-DOMAIN16 (LBD16) und LBD29 zur Regulierung der lateralen Wurzelorganogenese (Cho et al., 2014). BR spielt jedoch während dieses Prozesses eine untergeordnete Rolle und BIN2 steht unter der Kontrolle des TRACHEARY ELEMENT DIFFERENTIATION INHIBITORY FACTOR (TDIF)–TDIF RECEPTOR (TDR) Module (Cho et al., 2014). Zusammen können Sie Auxin-Reponsen regulieren, indem sie verschiedene Auxin-Signalkomponenten beeinflussen.,
Auxin reguliert die BR-Signalisierung
Andererseits kann Auxin in bestimmten Aspekten auch den BR-Signalweg regulieren. Die Expression von DWARF4, einer entscheidenden Hydroxylase für die BR-Biosynthese zur Kontrolle des endogenen BR-Spiegels, ist Auxin-abhängig. Die Auxin-Behandlung könnte die Expression von DWARF4 merklich stimulieren und Auxin könnte die Bindung von BZR1 an den Promotor von DWARF4 hemmen. Die Induktion von DWARF4 durch Auxin erfordert einen Auxin-Signalweg, jedoch keinen BR-Signalweg (Chung et al., 2011; Yoshimitsu et al., 2011)., CPD-katalysierende C-3-Oxidation von BR wurde durch BRX aktiviert, einen mutmaßlichen Transkriptionsfaktor, der stromabwärts der Auxin-Signalisierung wirkt (Mouchel et al., 2006). Weitere Studien in Reis zeigen, dass exogenes Auxin die Transkriptionsexpressionsniveaus des BR-Rezeptorgens OsBRI1 erhöhen kann, was darauf hindeutet, dass Auxin die BR-Signalisierung durch die Regulation von BR-Rezeptoren verstärkt (Sakamoto et al., 2013). Des Weiteren besitzt der Promotor von OsBRI1 ein Upstream-Auxin-Response-Element (AuxRE) – Motiv, das durch ARF-Transkriptionsfaktoren gezielt wird., Darüber hinaus zeigen Mutantenstudien, dass bei der Mutation von AuxRE die Induktion der Expression von OsBRI1 durch Auxin abgeschafft wird und auch die Expression von OsBRI1 in der arf-Mutante down reguliert wird (Sakamoto et al., 2013). Es wurde berichtet, dass OsARF19 an den Promotor von OsBRI1 bindet und seine Expression positiv reguliert, was dann die BR-Signalisierung aktiviert (Zhang et al., 2015). BES1 kann an den Promotor der KLEINEN AUXIN-UP-RNA 15 (SAUR15) binden und ein frühes Ansprechgen in Arabidopsis vermitteln, und diese Bindung könnte durch Auxin-Behandlung verbessert werden (Walcher und Nemhauser, 2012)., Zusammengenommen kann Auxin auch das Wachstum und die Entwicklung von BR-Reaktionen und BR-regulierten Pflanzen beeinflussen.
Abschließende Bemerkungen und Zukunftsperspektive
In den letzten fast vier Jahrzehnten haben Studien zu Auxin-BR-Wechselwirkungen immer mehr Interesse der Forscher geweckt. Die Verwendung physiologischer, molekularer, genetischer und biochemischer Werkzeuge hat unser Verständnis dieses Problems erheblich vertieft., Basierend auf den vorherigen Studien sind BR und Auxin synergistisch an mehreren Pflanzenentwicklungsprozessen beteiligt, darunter: Hypocotyldehnung, Entwicklung von Gefäßbündeln, Wurzelentwicklung und Tropismen usw. Die gegenseitige Abhängigkeit und Zusammenarbeit von Auxin und BR ist kompliziert und umfasst zahlreiche Prozesse auf molekularer Ebene, indem dieselben Zielgene auf mehreren Ebenen gegenseitig reguliert werden (Abbildung 1).
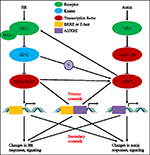
ABBILDUNG 1. Modell von auxin-brassinosteroid (BR) übersprechen., In der Arabidopsis werden die Wahrnehmungen des BR-und Auxin-Signals von BRI1-bzw. BR bindet an die extrazelluläre Domäne von BRI1 und fördert, dass es mit Co-Rezeptor BAK1 interagiert, um einen aktiveren BR-Rezeptorkomplex zu bilden, der wiederum zur Dephosphorylierung und Inaktivierung von BIN2 führt. Die Inaktivierung von BIN2 führt zur Dephosphorylierung von zwei BR-homologen Transkriptionsfaktoren BZR1 und BZR2, die sich in den Kern bewegen, um die Transkription von BRRE-oder E-Box-haltigen Genen in ihrer Promotorregion zu aktivieren., BIN2 kann auch ARF7 und ARF19 phosphorylieren, um ihre Wechselwirkung mit AUX/IaaS zu unterdrücken und dadurch die Transkriptionsaktivität ihrer Zielgene zu verbessern. TIR1 aktiviert die Auxin-Signalisierung und interagiert mit AUX/IAA-Proteinen als Co-Rezeptor von Auxin. Die AUX / IAA wird dann durch den Ubiquitin–Proteasom-Weg abgebaut, und die Auxin-Transkriptionsregler Auxin Response Factors (ARFs) werden aus der AUX/IAA-Repression freigesetzt und aktivieren die Transkription von Genen mit Auxin Responsive Elements (AUXRE) in ihrem regulatorischen Bereich., Einige ARFs können sich auch an den Promotor von BRI1 binden und seine Expression positiv regulieren, wodurch dann die BR-Signalisierung aktiviert wird. Der primäre Übersprechen erfolgt durch Aktivierung von Genen, die sowohl BRRE/E-Box als auch AUXRE in ihrer Promotorregion enthalten, so dass beide Signalwege die Transkription direkt regulieren können. Sekundärer Übersprechen erfolgt durch Expression von Genen, die entweder Auxin oder BR reagieren, aber deren Aktivitäten steuern Expression von Genen, die die Reaktion und Signalisierung anderer Hormone regulieren.,
Die Phosphorylierungsregulation spielt eine entscheidende Rolle für den BR-Signalweg, insbesondere während des Wahrnehmungsprozesses wird BR durch BRI1-Kinase-Rezeptor und BAK1-Kinase-Co-Rezeptoren wahrgenommen und steuert schließlich die BR-regulierte Genexpression durch Beeinflussung nachgeschalteter Transkriptionsfaktoren wie BES1 / BZR1-Aktivitäten (He et al., 2005; Yin et al., 2005; Sun et al., 2010; Tang et al., 2011; Yu et al., 2011). Die Ubiquitinationsregulierung scheint jedoch für die Auxin-Signalisierung unerlässlich zu sein., Sobald Auxin an den TIR1-Rezeptor bindet, der als Ubiquitin E3-Ligase wirkt, ubiquitiniert die aktivierte TIR1 E3-Ligase AUX/IAA-Proteine, führt zum Abbau dieser Repressoren und unterdrückt ARF-Transkriptionsfaktoren und verursacht schließlich Veränderungen des Auxin-regulierten Genexpressionsmusters und Wachstumsreaktionen (Gray et al., 1999, 2002; Hellmann et al., 2003; Quint et al., 2005). Da festgestellt wurde, dass die BIN2-Kinase, von der bekannt ist, dass sie bei der BR-Signalisierung funktioniert, die Aktivitäten von ARFs wie ARF2 und ARF7 phosphorylieren und verstärken könnte (Vert et al., 2008; Cho et al.,, 2014) wird es interessant sein zu testen, ob Kinasen wie BIN2, die an der BR-Signalisierung beteiligt sind, auch mit anderen Auxin-Signalkomponenten wie TIR1-Rezeptor-oder AUX/IAA-Repressoren interagieren und TIR1 E3-Ligase-Aktivität oder AUX/IAA-Proteinstabilitäten beeinflussen können. Andererseits muss auch die Rolle der Ubiquitination bei der BR-Signalisierung angesprochen werden, insbesondere wenn TIR1 E3-Ligase direkt mit BR-Signalkomponenten interagieren und ihre Proteinstabilitäten regulieren könnte.,
Darüber hinaus wurde unter Verwendung von Auxin Response DR5 und anderen Auxin-Reportern beobachtet, dass Auxin das Pflanzenwachstum und die Pflanzenentwicklung in gewebeabhängiger oder zellulärer Weise reguliert. Die unterschiedlichen Transkriptionsausgaben hängen vom zellulären und ökologischen Kontext ab (Clark et al., 2014; Etchells et al., 2016; Lavy et al., 2016)., Obwohl gezeigt wurde, dass die räumlich-zeitliche BR-Signalisierung das Wurzelwachstum durch die antagonistische Wirkung mit Auxin steuert (Chaiwanon und Wang, 2015), ist es immer noch unbekannt, ob die Gewebe-oder zelluläre BR-Signalisierung, die durch pBZR1:BZR1-YFP visualisiert werden könnte, auch wichtig ist, um andere Prozesse außer der Wurzelentwicklung zu kontrollieren. Weiterhin ist derzeit die Erzeugung einer detaillierten Gewebe-oder Zellkarte von Auxin-und BR-Verteilungen mittels fluoreszenzaktivierter Zellsortierung oder Lasermikrodissektion in Kombination mit hochauflösender Genexpressionsanalyse möglich., Dies führt schließlich zu adressieren, wenn sich das Auxin mit BR in einer Gewebe-oder zellulären Weise kreuzt.
Autor Beiträge
Alle Autoren beteiligt waren, in das schreiben dieser rezension Manuskript. Weitere Informationen darüber, was Urheberschaft ausmacht, finden Sie in unseren Autorenrichtlinien.,
Finanzierung
Diese Arbeit wird durch Zuschüsse der National Natural Science Foundation of China (Projekte 31500229 und 31770305), der Shandong Province Natural Science Foundation des Major Basic Research Program (2017C03) und der Shandong Provincial Funds for Distinguished Young Scholars (2014JQ201408) unterstützt.
Interessenkonflikterklärung
Die Autoren erklären, dass die Untersuchung ohne kommerzielle oder finanzielle Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Clouse, S. D. (2011)., Brassinosteroid-Signaltransduktion: von der Aktivierung der Rezeptorkinase bis zu transkriptionellen Netzwerken, die die Pflanzenentwicklung regulieren. Pflanzenzelle 23, 1219-1230. doi: 10.1105/tpc.111.084475
PubMed Abstract | CrossRef Full Text | Google Scholar
Estelle, M. (2011). Auxin Signalisierung: Von der Synthese zur Systembiologie; A Subject Collection from Cold Spring Harbor Perspectives in Biology. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press.
Google Scholar
Guilfoyle, T. J. (2015)., The PB1 domain in auxin response factor and Aux/IAA proteins: a versatile protein interaction module in the auxin response. Plant Cell 27, 33–43. doi: 10.1105/tpc.114.132753
PubMed Abstract | CrossRef Full Text | Google Scholar