Introduzione
La mortalità dei suinetti è uno dei principali tratti di selezione nell’allevamento dei suini ed è influenzata dalla scrofa, dai suinetti e dall’ambiente. Quindi, la mortalità dei suinetti è un fenotipo complesso e dipende dalla capacità della scrofa di allevare la sua prole, ma è anche una funzione del peso alla nascita, della gestione e della selezione (Knol et al., 2002)., Tuttavia, anche i difetti recessivi monogenici contribuiscono alla mortalità dei suinetti, anche se solo pochi esempi sono stati riportati in passato (Murgiano et al., 2012; Matika et al., 2019). Anche in quei casi in cui l’effetto della mutazione è grave, la selezione efficiente contro tale mutazione è ostacolata dalla bassa frequenza. In molti difetti gravi, gli zigoti muoiono molto presto nella gestazione, senza lasciare traccia se non l’assenza di omozigoti nella popolazione in generale (Derks et al., 2019).,
Gli effetti Inbreeding nelle popolazioni di suini commerciali sono solitamente tenuti sotto controllo dall’allevamento selettivo per una diminuzione della mortalità nei suinetti migliorando sia le capacità materne che la vitalità dei suinetti (Olijslagers, 2018). Tuttavia, le varianti alla base dei difetti monogenici recessivi non sono ben catturate all’interno dei valori di allevamento e potenzialmente vanno alla deriva a frequenze più alte come risultato di un’intensa selezione (Georges et al., 2019). Inoltre, queste varianti possono anche essere mantenute come risultato della selezione di bilanciamento per un effetto positivo correlato in stato eterozigote (Derks et al., 2018).,
I difetti recessivi contribuiscono solo marginalmente alla mortalità complessiva dei suinetti (Alonso-Spilsbury et al., 2007). Tuttavia, le varianti che influenzano la mortalità dei suinetti sono di grande importanza perché queste varianti influenzano direttamente la produzione e il benessere degli animali (Baxter et al., 2013; Rutherford et al., 2013). Tuttavia, nella gestione della popolazione animale, l’insorgenza di difetti a bassa frequenza è solitamente scarsamente documentata (spesso vengono utilizzati termini molto generali) e le sindromi sono spesso riconosciute solo una volta raggiunta un’alta frequenza., Questo è particolarmente rilevante per le sindromi che non portano a fenotipi molto distinti. Pertanto, anche nelle popolazioni riproduttrici commerciali si può fare poco monitoraggio su specifiche sindromi, e selezionare efficacemente contro specifiche sindromi a bassa frequenza richiede quindi nuovi approcci.
In questo lavoro, descriviamo la scoperta di una sindrome altamente debilitante in una popolazione di suini commerciali attraverso un sondaggio basato su un array SNP combinato a media densità e sequenziamento dell’intero genoma (WGS)., Il sondaggio ha portato all’identificazione di una delezione di frameshift 16-bp nel gene SPTBN4, con chiare conseguenze fenotipiche previste negli omozigoti. La frequenza portante è di circa il 9% nella popolazione in studio, interessando circa lo 0,81% delle cucciolate della popolazione. La frequenza era sufficientemente bassa da essere sconosciuta per avere una base genetica e persino essere effettivamente non riconosciuta come una sindrome specifica. Al momento dell’attuazione del sondaggio, una scrofa incinta è stata identificata da un cinghiale portatore., I suinetti colpiti soffrono di miopatia e non sono in grado di camminare, di solito con conseguente morte entro poche ore dopo la nascita, completamente in linea con la patologia prevista rispetto a casi simili umani e topi.
Materiale e metodi
Animali, genotipi e pre-elaborazione
Il set di dati è costituito da 31.839 animali provenienti da una linea sintetica di cinghiali con ampio sfondo bianco. La linea viene mantenuta e allevata nelle fattorie Topigs Norsvin nucleus, selezionando principalmente i tratti di produzione e salute., Gli animali sono stati genotipizzati sul chip Illumina GeneSeek custom 50K SNP (Lincoln, NE, USA). Animali con una frequenza di genotipi mancanti > 0,15 sono stati rimossi. Abbiamo scartato i marcatori che non soddisfacevano i seguenti criteri di filtraggio: una frequenza di chiamata minima di 0,85, una frequenza allele minore> 0,01 e un valore p-test esatto delle proporzioni Hardy-Weinberg inferiore a P< 10-12. Inoltre, i marcatori con posizione sconosciuta sulla build del genoma Sscrofa11.1 sono stati scartati, lasciando 41.573 marcatori dopo il filtraggio., Tutti i passaggi sono stati eseguiti in Plink v1.90b3 .30 (Purcell et al., 2007).
Fasatura dell’aplotipo e identificazione dell’aplotipo SSC6
Abbiamo eseguito fasatura dell’aplotipo e imputazione dei siti mancanti in Beagle5.0 con parametro per dimensione effettiva della popolazione impostato su 100, altre impostazioni erano predefinite (Browning et al., 2018). Gli omozigoti attesi sono stati stimati in base alla frequenza dell’aplotipo, utilizzando il principio di Hardy-Weinberg. È stato applicato un test binomiale esatto per testare il numero di omozigoti osservati con il numero di omozigoti attesi., L’aplotipo è stato considerato significativamente impoverito se P < 5 × 10-3.
Effetti fenotipici associati all’aplotipo SSC6
Abbiamo esaminato l’aplotipo SSC6 per i record sul numero totale nato, numero nato morto, suinetti mummificati, sopravvivenza da parto e sopravvivenza all’allattamento (sopravvivenza fino a circa 21 giorni di età) su un totale di 9.666 cucciolate. Abbiamo elencato questi fenotipi per tutte le cucciolate CXC e CXN identificate. Abbiamo usato un t-test di Welch per valutare se i fenotipi delle cucciolate CxC differiscono significativamente dalle cucciolate CxN. Un valore p < 0.,05 è stato considerato significativo.
Analisi di sequenziamento del genoma intero e identificazione della variante candidata
Il set di dati è costituito da 71 individui sequenziati del genoma intero della popolazione in studio. Tutti i 71 campioni erano presenti anche nel nostro set di dati di 31.839 animali genotipizzati sul 50K. I 71 campioni hanno un volume totale di 1,93 Tbp (coppie di basi tera) da 14,16 miliardi di letture accoppiate a 150 bp (Tabella S3). I campioni sono stati sequenziati su Illumina HiSeq 2000. Abbiamo allineato le sequenze al genoma Sscrofa11. 1 costruire utilizzando BWA-MEM versione 0.7.,15 (Li e Durbin, 2009) con una mappabilità media del 98,9% e una copertura del campione che varia da 8,8 a 14,8 X (media 10,9 X). Samblaster è stato utilizzato per rimuovere i duplicati PCR (Faust e Hall, 2014). Samtools è stato utilizzato per ordinare, unire e indicizzare i file bam (Li et al., 2009). Le statistiche di mappatura e qualità sono state generate utilizzando Qualimap (Okonechnikov et al., 2016). La chiamata variante è stata eseguita con Freebayes v1.1.0 con le seguenti impostazioni: –min-base-quality 10 –min-alternate-fraction 0.2 –haplotype-length 0 –min-alternate-count 2 (Garrison and Marth, 2012)., Varianti con punteggio di qualità Phred < 20 sono state scartate (Li et al., 2009). Le varianti sono state annotate usando l’Ensembl variant effect predictor (VEP, release 96) (Mclaren et al., 2016). L’impatto delle varianti missense è stato previsto usando l’ordinamento intollerante da tollerante (SIFT) (Kumar et al., 2009). L’analisi LD è stata eseguita utilizzando Plink v1.90b3.30 (Purcell et al., 2007) con le seguenti impostazioni-chr-set 18, – r2, ld-window-r2 0.8.
Allineamento delle proteine SPTBN4
L’allineamento delle proteine tra il tipo selvaggio e la proteina mutante è stato eseguito utilizzando ClustalO (Madeira et al.,, 2019) e visualizzato utilizzando ESPript 3 (Robert e Gouet, 2014). Ulteriore visualizzazione e convalida è stata eseguita utilizzando la versione jbrowse genome viewer 1.12.1 (Skinner et al., 2009).
La convalida della delezione causale 16 bp SPTBN4
PCR è stata eseguita utilizzando 60 ng di DNA genomico, con 0,4 µm di ciascun primer, 1,8 mm MgCl2 e 25 unità / ml di OneTaq® DNA Polimerasi (OneTaq® 2X Master Mix con tampone Standard, New England Biolabs) nel tampone PCR del produttore in un volume finale di 12 µl., Denaturazione iniziale per 1 min a 95°C è stata seguita da 35 cicli di 95 ° C per 30 s, 55°C per 45 s, 72°C 90 s, seguita da un 5 min estensione 72°C. primer PCR per SPTBN4 sono TCAAGGGTGCAGGCTCTTTC avanti e GGTAGGAAGCTCGAAGTGGG reverse. Il primer forward è stato etichettato con colorante con 6-FAM per produrre un prodotto PCR etichettato in modo fluorescente rilevabile su ABI 3730 DNA sequencer (Applied Biosystems). Le dimensioni dei frammenti sono state determinate utilizzando il software GeneMapper 5 di ABI.,
Esame istopatologico
Due suinetti affetti di età inferiore a 1 settimana sono stati inviati al dipartimento di patologia della Royal Animal Health (Deventer) per l’esame. Macroscopicamente, tutte le osservazioni erano entro i limiti normali. Il muscolo scheletrico della zampa anteriore, il muscolo dorsale e la gamba posteriore di entrambi gli animali sono stati campionati per la colorazione di routine H&E e la colorazione PTAH. Il tessuto muscolare è stato conservato in vasetti separati e fissato in soluzione di formaldeide 4%, tamponata (=soluzione di formalina 10%, tamponata)., Successivamente, il tessuto è stato incorporato in paraffina e tagliato in 2 µm secondo la procedura operativa standard (SOP RAH). Successivamente, i vetrini sono stati deparaffinizzati e regolarmente colorati per ematossilina ed eosina (H&E) in una macchina automatica a colori. Contemporaneamente sono stati preparati vetrini aggiuntivi di 2 µm del tessuto muscolare e un vetrino di controllo positivo del tessuto muscolare per la colorazione manuale con “ematossilina dell’acido fosfotungstico”, abbreviato in PTAH. Questa colorazione è preferita per dimostrare le striature incrociate del muscolo scheletrico.,
Valori di allevamento e analisi di associazione
In questo studio, abbiamo valutato 63 tratti utilizzati nel programma di allevamento. Come variabile di risposta per ogni tratto in studio sono stati utilizzati valori di allevamento stimati deregressi (DEBV). Il valore di allevamento stimato (EBV) di tutti i tratti valutati è stato deregresso utilizzando la metodologia descritta da Garrick et al. (2009). L’EBV di ciascun animale è stato ottenuto dalla valutazione genetica di routine da parte di un programma di allevamento commerciale (Topigs Norsvin) utilizzando un modello animale., Le affidabilità per animale ai fini della deregressione sono state estratte dalla valutazione genetica basata sulla metodologia di Tier e Meyer (2004). Le eredità utilizzate per la deregressione sono state estratte anche dalla valutazione genetica di routine. Infine, i fattori di ponderazione basati sull’affidabilità stimata del DEBV sono stati stimati anche secondo Garrick et al. (2009) utilizzando un valore di 0,5 per lo scalare c. Per garantire la qualità del DEBV, solo gli animali con un fattore di ponderazione maggiore di zero e un’affidabilità del DEBV maggiore di 0.,20 sono stati utilizzati nelle analisi di associazione. L’affidabilità del DEBV è stata ottenuta anche secondo Garrick et al. (2009).
Le analisi di associazione sono state eseguite utilizzando il software ASREML (Gilmour et al.,, 2009) l’applicazione lineare misto modello animale:
dove DEBVij è osservato DEBV per l’animale j, w è il fattore di ponderazione per il residuo, µ è la complessiva DEBV media della popolazione, Ri è il vettore di stato (conte di negativo allele) del 4 mutazione io, aj è l’effetto genetico additivo stimato con un pedigree-in base al rapporto medio matrice, e l’errore residuo. Le associazioni con a-log10(valore P) maggiore di cinque sono state dichiarate significative.
Risultati
A 1.,Il segmento 5 Mb sul cromosoma 6 influisce sulla sopravvivenza dell’allattamento nei suini
Abbiamo analizzato 31.638 animali da una singola linea di cinghiale di razza pura (linea sintetica con grande sfondo bianco), genotipizzata sul chip SNP 50K suino (build Sscrofa11.1) (Warr et al., 2019). L’analisi ha rivelato un segmento di 1,5 Mb sul cromosoma 6 (SSC6:48,75–50,25) che mostra un deficit di omozigosità associato a una ridotta sopravvivenza alla lattazione (Tabelle 1 e 2). L’aplotipo si segrega con una frequenza allelica moderata del 4,5% (9,0% di frequenza portante) nella popolazione in studio., La frequenza dell’aplotipo è stata fluttuante nell’ultimo decennio, ma è diminuita negli ultimi 3 anni (Figura S1). Abbiamo testato se la frequenza era guidata da un effetto vantaggio eterozigote. Tuttavia, abbiamo trovato associazioni per lo più negative con importanti tratti di selezione ad eccezione della profondità del lombo e della lunghezza della gestazione (Tabella 3), il che suggerisce che la frequenza è puramente il risultato della deriva genetica.

Tabella 1 Caratteristiche dell’aplotipo SSC6.,

Tabella 2 Le cucciolate carrier-by-carrier mostrano una diminuzione del 24% nella sopravvivenza dell’allattamento rispetto alle cucciolate carrier-by-carrier. I risultati significativi sono indicati in grassetto.

Tabella 3 Tratti significativamente associati ai portatori eterozigoti della cancellazione SPTBN4.
Le 52 cucciolate carrier-by-carrier (CxC) non mostrano una riduzione significativa del numero totale di animali nati o nati vivi., Tuttavia, la sopravvivenza all’allattamento è ridotta di circa il 24% nelle cucciolate CxC rispetto agli accoppiamenti carrier-by-noncarrier (CxN), indicando che i suinetti omozigoti muoiono entro il periodo di allattamento (Tabella 2). Successivamente, abbiamo esaminato le osservazioni per il tempo e la causa della mortalità delle cucciolate CxC. Questo ha rivelato che la maggior parte dei suinetti che sono morti entro il primo 24 h dopo la nascita. La maggior parte di questi suinetti sono stati per lo più descritti dagli agricoltori come “maialino debole alla nascita.,”
L’analisi di sequenziamento dell’intero genoma rivela una delezione di frameshift 16-bp in SPTBN4 come probabile variante causale
Per identificare la mutazione causale, abbiamo esaminato i dati di sequenza dell’intero genoma da 71 animali della popolazione in studio e identificato cinque animali portatori. L’analisi del disequilibrio del legame (LD) ha rivelato 267 varianti SNP e indel in alto LD (r2 > 0.8) con l’aplotipo SSC6 (Tabella S1), la maggior parte in perfetto LD (247 varianti)., Solo cinque varianti influenzano potenzialmente la sequenza di codifica (tre missense, un frameshift, un accettore di giuntura). Si prevede che le tre varianti missense siano tollerate da SIFT (score > 0.18, Tabella S1), mentre la variante splice-accettore colpisce un gene che codifica un peptide a 28 bp di funzione sconosciuta, improbabile che sia causale. Tuttavia, è stato previsto che una variante in LD completo (r2 = 1) con l’aplotipo abbia un impatto elevato; una delezione di frameshift di 16 bp nell’esone 26 del gene SPTBN4 (6:g.48801280delGACGGTGTACGCCGGT) (Figure 1A, B)., La delezione del frameshift (ENSSSCP00000031537:p.Arg1902fs) introduce 30 nuovi aminoacidi e un codone di arresto prematuro, producendo una proteina beta non eritrocitaria 4 (SPTBN4) alterata e troncata di spettrina. I mutanti mancano dei 662 amminoacidi finali della proteina wild type (Figura 1C), incluso il dominio di omologia pleckstrin (PH) richiesto per il trasporto delle proteine alle membrane (Wang et al., 2018). La proteina SPTBN4 è un membro delle proteine beta-spettrina ed è un’actina che collega la membrana cellulare al citoscheletro di actina., Le mutazioni di SPTBN4 interrompono il meccanismo citoscheletrico che controlla la corretta localizzazione dei canali ionici nei nervi mielinizzati causando neuropatie motorie (Parkinson et al., 2001; Wang et al., 2018).

Figura 1 (A) Modello genico SPTBN4. La posizione del 26 ° esone interessato è indicata in rosso. (B) Illustrazione della cancellazione 16-bp. La figura mostra il tipo selvaggio e l’esone mutante. (C) Allineamento della sequenza proteica mutante (Mt) e wild type (Wt) SPTBN4. La mutazione induce 30 nuovi amminoacidi e un codone di arresto prematuro.,
La genotipizzazione di cinque cucciolate CxC conferma la delezione SPTBN4 come probabile colpevole
Abbiamo genotipizzato cinque cucciolate CxC per la delezione a 16 bp che aveva almeno due suinetti (intervallo 2-6) morti entro le prime 48 ore dopo la nascita. Le cinque cucciolate hanno prodotto 53 suinetti di cui 19 omozigoti per la delezione di 16 bp (Tabella 4). Tutti i suinetti omozigoti 19 sono morti entro 48 h dopo la nascita (18 entro 24 h). Dei restanti 34 suinetti (8 wild type e 26 portatori), solo 1 è morto entro 48 h, probabilmente causato da altri fattori (ambientali).,

Tabella 4 Genotipizzazione della probabile cancellazione del frameshift 16-bp SPTBN4 causale in cinque cucciolate carrier-by-carrier. La somma per classe di genotipo è indicata in grassetto.
I suinetti omozigoti per la delezione SPTBN4 soffrono di miopatia e paralisi degli arti posteriori
Abbiamo monitorato una recente cucciolata CxC (data di parto: 28 aprile 2019) che ha prodotto sei sani, due affetti (campioni: 9912, 9916) (Figura 2A) e tre suinetti nati morti., Abbiamo confermato lo stato omozigote di eliminazione di SPTBN4 per i due suinetti colpiti (Tabella S2). Inoltre, abbiamo osservato quattro portatori eterozigoti e due suinetti omozigoti di tipo selvatico tra gli individui sani. Uno dei suinetti nati morti (campione: 9921) era anche omozigote per la delezione, mentre gli altri due erano eterozigoti. I suinetti colpiti soffrono di estrema debolezza muscolare (Figure 2B, C), paralisi degli arti posteriori e tremori (video S1). Quindi, i maialini non erano in grado di camminare o bere.,
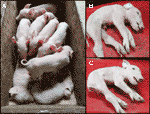
Figura 2 (A) Due suinetti colpiti (vivi) insieme a sei littermates sani. I suinetti derivano da un accoppiamento CxC partorito il 28 aprile 2019. B) Maialino maschio interessato 9912. C) Suinetti femmina colpiti 9916.
I suinetti affetti mancano di striature incrociate nei muscoli scheletrici degli arti dorsali e posteriori
L’esame istopatologico ha rivelato degenerazione sparsa delle fibre muscolari in entrambi i suinetti e necrosi e vasculite focalmente nel muscolo dorsale in uno dei suinetti (ID = 9912)., Inoltre, la colorazione dell’ematossilina dell’acido fosfotungstico (PTAH) mostra una colorazione divergente delle fibre muscolari scheletriche, indicando una diminuzione delle striature incrociate, in particolare nei muscoli delle zampe dorsali e posteriori degli animali colpiti (Figura 3B), mentre le zampe anteriori sembrano inalterate (Figura 3A). La diminuzione delle striature incrociate è indicata dalla colorazione anormale e dalla perdita generale di volume delle fibre muscolari (Figura 3B). I cambiamenti istopatologicamente osservati nelle zampe posteriori e nei muscoli dorsali sono indicativi di distrofia muscolare.,

Figura 3 (A) Vista in sezione trasversale di un muscolo scheletrico dalla gamba anteriore. La freccia nera indica la colorazione normale (scura) delle fibre muscolari che indica la presenza di striature incrociate. PTAH Bar = 50 µm. (B) Vista in sezione trasversale di un muscolo scheletrico dalla gamba posteriore. La freccia nera indica una colorazione anormale (rosa) delle fibre muscolari che indica la mancanza di striature incrociate. La freccia gialla indica la colorazione normale e la presenza di striature incrociate. PTAH Bar = 50 µm.,
Discussione
In questo lavoro riportiamo un nuovo difetto congenito che causa la mortalità dei maialini probabilmente a causa di una delezione del frameshift di 16 bp nel gene SPTBN4. I suinetti soffrono di estrema debolezza muscolare (miopatia) e muoiono entro poche ore dalla nascita. Si prevede che la delezione conferisca una completa perdita di funzione della beta spettrina, proteina 4 non eritrocitaria. SPTBN4 è un membro della famiglia dei geni della spettrina ed è richiesto per il clustering dei canali ionici nei nodi di Ranvier, influenzando il potenziale d’azione (Devaux, 2010)., Le mutazioni interrompono il meccanismo citoscheletrico che controlla la corretta localizzazione dei canali ionici e la funzione dei domini assonali principalmente nei segmenti iniziali dell’assone (AIS) e nei nodi di Ranvier (Wang et al., 2018). Più specificamente, il dominio C-terminale interessato di SPTBN4 è cruciale per il traffico del canale KCNQ2 e l’eccitabilità ai nodi di Ranvier (Devaux, 2010).
La successiva ricerca di follow-up ha identificato casi umani e di topo che indicavano che la sindrome che ne derivava probabilmente non si sarebbe dimostrata immediatamente letale, ma piuttosto conferiva una grave miopatia., Con i dati di genotipo SNP a media densità, disponibili per tutti gli animali della popolazione riproduttiva (N = 31.839), è stato possibile identificare i portatori. Tra questi portatori c’era una scrofa che era approssimativamente a medio termine in gravidanza al momento dell’identificazione, generata da un cinghiale che era anche portatore. L’allevamento è stato notificato per documentare la cucciolata alla nascita. Il fenotipo osservato dei suinetti colpiti (miopatia, paralisi degli arti posteriori, tremori) era completamente congruente con quanto osservato nei pazienti umani con perdita omozigote della funzione o mutazioni eterozigoti composte nel gene SPTBN4 (OMIM: 606214)., Due dei pazienti umani hanno mutazioni di perdita di funzione all’interno del dominio del PH (Wang et al., 2018), sostenendo che una perdita del dominio del PH nei suini porterebbe probabilmente a una completa perdita di funzione della proteina SPTBN4. Nell’uomo, mutazioni simili portano a una grave miopatia congenita causata dall’assenza di fibre muscolari di tipo I, neuropatia e sordità (Knierim et al., 2017; Wang et al., 2018). Wang et al. (2018) ha anche osservato neuropatia assonale motoria in diversi pazienti caratterizzati da ipotonia congenita, profonda debolezza e perdita di riflessi tendinei profondi dalla prima infanzia., Inoltre, le biopsie nervose hanno rivelato canali Na + nodali ridotti e nessun canale KCNQ2 K+ nodale, rivelando la patologia molecolare che causa disfunzione del sistema nervoso. Pertanto, concludiamo che questa variante di frameshift è la probabile mutazione causale che porta al fenotipo osservato e all’esaurimento del genotipo omozigote nella popolazione. Studi futuri potrebbero concentrarsi sulla realizzazione di un knockout in vivo del gene SPTBN4 nel maiale, per studiare la sindrome e il fenotipo associato in modo più dettagliato.,
Non abbiamo osservato la degenerazione delle fibre muscolari nelle zampe anteriori, mentre le fibre muscolari dorsali e posteriori erano chiaramente colpite. Questa osservazione potrebbe in parte spiegare la paralisi degli arti posteriori, mentre le zampe anteriori non sono interessate. La discrepanza tra le fibre muscolari delle zampe anteriori e posteriori è stata descritta anche nei topi tremanti, in cui le mutazioni SPTBN4 loss-off-function causano neuropatia motoria, paralisi degli arti posteriori, tremori e sordità centrale (Parkinson et al., 2001; Komada e Soriano, 2002). Parkinson et al., (2001) descrivono ridotte velocità di conduzione nervosa nei nervi sciatici di topi con alleli tremanti che causano la neuropatia periferica degli arti posteriori. L’espressione di SPTBN4 nei topi è limitata al cervello, al midollo spinale e ai nervi sciatici e non osservata nel muscolo scheletrico, quindi questa malattia è principalmente un difetto neuronale. Nel complesso non è chiaro quale meccanismo causi l’assenza di sintomi negli arti anteriori. Questo “knockout naturale” nei suini può essere una risorsa utile per studiare la malattia umana, poiché i suini sono di solito un modello migliore per studiare la malattia umana rispetto alle specie di roditori., Inoltre, la conseguenza della perdita della funzione SPTBN4 può essere studiata in modo più dettagliato.
La dimensione effettiva della popolazione (Ne) della razza in studio è stimata intorno a 100 (Hidalgo et al., 2016). Nell’allevamento degli animali, un basso Ne aumenta il rischio che gli alleli dannosi aumentino di frequenza per caso. Inoltre, studi precedenti hanno dimostrato che gli alleli letali recessivi possono essere guidati da effetti vantaggiosi negli eterozigoti (Derks et al., 2018; Matika et al., 2019). Matika et al.,, 2019 ha trovato una mutazione recessiva stop-gained nel gene MSTN associato ad un importante aumento della profondità muscolare negli eterozigoti. Tuttavia, non troviamo prove di alcun vantaggio eterozigote nel nostro studio. Con le attuali tecniche genomiche possiamo ora identificare alleli deleteri che vanno alla deriva a frequenze più alte e monitorare l’emergere di nuovi alleli deleteri con precisione, consentendo uno spurgo più efficace. Inoltre, il risultato di questo tipo di studio migliorerà notevolmente la coscienza dei difetti genetici “nascosti” sia a livello di allevatore che di agricoltore., Senza alcuna informazione preventiva, difetti alla nascita rari sono spesso registrati come ” maialino debole.”E senza alcuna ulteriore distinzione di sindromi specifiche, ulteriori azioni non sono possibili. Nella maggior parte dei casi non è noto se esiste una base genetica o che potrebbero esserci altri effetti confondenti. Con informazioni genomiche precedenti, la sindrome può essere identificata, rispetto ad altri casi, e i portatori identificati, portando a informazioni attuabili.
La mortalità dei suinetti è di grande importanza economica e per il benessere degli animali., Quindi, la scoperta della mutazione SPTBN4 ha portato all’implementazione immediata nel programma di allevamento per ridurre al minimo la frequenza degli accoppiamenti carrier-by-carrier. Ciò consente di evitare la nascita di individui affetti, migliorando così il benessere degli animali e riducendo le perdite economiche.
Conclusione
In questo studio riportiamo un nuovo difetto congenito probabilmente causato da una delezione recessiva del frameshift nel gene SPTBN4 nei suini. I risultati sono supportati da sorprendenti somiglianze con i fenotipi sindromici associati a SPTBN4 negli esseri umani e nei topi., Lo studio consente di monitorare e spurgare l’allele deleterio dalla popolazione. Le croci vettore per vettore possono essere prevenute, precludendo le persone colpite, riducendo così le perdite economiche e migliorando il benessere degli animali. Infine, questi “knockout naturali” ottenuti nell’industria riproduttiva possono fornire un modello per la malattia umana e aumentare la nostra comprensione della funzione genica all’interno del clade dei mammiferi e fornire un potenziale modello per la malattia umana.,
Dichiarazione sulla disponibilità dei dati
Dichiarazione sull’etica
La revisione e l’approvazione etica non sono state richieste per lo studio sugli animali perché i dati utilizzati in questo studio sono stati ottenuti come parte della raccolta di dati di routine dai programmi di allevamento Topigs Norsvin e non specificamente ai fini di questo progetto. Pertanto, l’approvazione di un comitato etico non era obbligatoria. La raccolta dei campioni e la registrazione dei dati sono state condotte rigorosamente secondo la legge olandese sulla protezione e il benessere degli animali (Gezondheids-en welzijnswet voor dieren)., Il consenso informato scritto è stato ottenuto dai proprietari per la partecipazione dei loro animali a questo studio.
Contributi dell’autore
MG, H-JM e MD hanno concepito e progettato lo studio. BH era responsabile dell’organizzazione generale e della comunicazione con Topigs Norsvin e gli agricoltori. MD e ML hanno eseguito l’analisi dei dati. BD e KL hanno eseguito lavori di laboratorio. SG-V ha eseguito l’analisi patologica. MD ha scritto il manoscritto. H-JM, MG, BH, SG-V, BD, KL e ML hanno fornito utili commenti e suggerimenti e hanno contribuito a redigere il manoscritto. I dati fenotipici sono stati analizzati mediante ML., Tutti gli autori hanno letto e approvato il manoscritto finale.
Finanziamento
Questa ricerca è stata finanziata dalla partnership STW-Breed4Food, progetto numero 14283: From sequence to phenotype: detecting deleterious variation by prediction of functionality. Questo studio è stato sostenuto finanziariamente da NWO-TTW e dai partner Breed4Food Cobb Europe, CRV, Hendrix Genetics e Topigs Norsvin. Inoltre, questo studio è stato supportato dal progetto IMAGE (Horizon 2020, n. 677353). I finanziatori non avevano alcun ruolo nella progettazione dello studio, nella raccolta e nell’analisi dei dati, nella decisione di pubblicare o nella preparazione del manoscritto., L’uso del cluster HPC è stato reso possibile da CATAgroFood (Strutture di ricerca condivise Wageningen UR).
Disclaimer
I dati utilizzati in questo studio sono stati ottenuti come parte della raccolta di dati di routine dai programmi di allevamento Topigs Norsvin, e non specificamente per lo scopo di questo progetto. Pertanto, l’approvazione di un comitato etico non era obbligatoria. La raccolta dei campioni e la registrazione dei dati sono state condotte rigorosamente secondo la legge olandese sulla protezione e il benessere degli animali (Gezondheids-en welzijnswet voor dieren).,
Conflitto di interessi
ML e BH sono dipendenti del Topigs Norsvin Research Center, un istituto di ricerca strettamente legato a uno dei finanziatori (Topigs Norsvin).
Tutti i restanti autori dichiarano che la ricerca è stata condotta in assenza di rapporti commerciali o finanziari che potrebbero essere interpretati come un potenziale conflitto di interessi.
Gli altri partner di Breed4Food Cobb Europe, CRV, Hendrix Genetics, dichiarano di non avere interessi concorrenti per questo studio.,
Ringraziamenti
Gli autori ringraziano Frank van Haaren e Toon Janssen per aver organizzato la comunicazione con la fattoria. Ringraziamo Manon Houben per il suo contributo agli esami patologici. Ringraziamo Mout Viller per aver scattato video e foto dei maialini colpiti e sani dopo la nascita. Ringraziamo Jürgen Harlizius per aver preso cura dei maialini dopo la consegna. Ringraziamo Gerda van Eldik per la raccolta dei campioni. Ringraziamo Egbert Knol e Egiel Hanenberg per il supporto generale e la guida di Topigs-Norsvin.,
Materiale supplementare
Video S1 / Video che mostra entrambi gli individui affetti dopo la nascita.
Tabella S1 / Variazione genomica in alta LD con l’aplotipo SSC6.
Tabella S2 / Genotipizzazione della cucciolata di CxC comprendente due individui affetti (data di parto: 28 aprile 2019). La tabella mostra lo stato omozigote del/del per i due suinetti colpiti 9912,9916.
Tabella S3 / Mappatura e statistiche di copertura da campioni WGS.
Figura S1 / SPTBN4 cancellazione frequenza portante dal 2007-2019.