Introduzione
A differenza degli animali che possono muoversi per evitare gli ambienti avversi, le piante sessili mostrano un adattamento altamente sviluppato alle complicate condizioni ambientali. Per raggiungere questa profonda adattabilità, sono necessarie comunicazioni tra le cellule. La comunicazione cella-cella nelle piante comporta una robusta elaborazione di segnalazione intracellulare e intricate reti di segnalazione intercellulari., Fino ad ora ci sono almeno nove sostanze di segnalazione, chiamate ormoni vegetali, tra cui auxina, brassinosteroide (BR), citochinina, gibberelline (GA), etilene, acido jasmonico (JA), strigolattone (SL), acido abscisico (ABA) e acido salicilico (SA) scoperti (Druege et al., 2016; Verma et al., 2016). Gli studi genetici e fisiologici hanno rivelato i ruoli critici e i meccanismi funzionali di questi ormoni di cui sopra nella crescita e nello sviluppo delle piante (Gray, 2004)., Sulla base degli studi precedenti, auxina, BR, GA, SL e citochinina funzionano principalmente durante la normale crescita e sviluppo delle piante, mentre ABA, etilene, JA e SA svolgono ruoli importanti nella risposta alla crescita delle piante a vari stress biotici e abiotici (Pieterse et al., 2009; Santner et al., 2009; Denance et al., 2013). E anche alcuni di questi ormoni hanno un doppio ruolo, ad esempio, l’ABA svolge anche ruoli importanti nello sviluppo dei semi e nella dormienza (Seo e Koshiba, 2002)., Sebbene ogni ormone svolga ruoli predominanti in determinati aspetti, molti ormoni hanno attività sovrapposte e le interazioni di diversi ormoni controllano molti aspetti dello sviluppo e della crescita in risposta a segnali di sviluppo ed esogeni endogeni.
Auxina e BR sono due classi principali di ormoni che promuovono la crescita., BR, un gruppo di ormoni steroidei specifici per le piante che potrebbero interagire con altri fitormoni come auxina, citochinina, etilene, GA, JA e SA e regolare una vasta gamma di processi di crescita e sviluppo delle piante, tra cui germinazione dei semi, allungamento delle cellule, differenziazione vascolare, formazione e movimento degli stomi, fioritura e fertilità maschile (Saini et al., 2015). È interessante notare che ciascuno di questi processi è anche controllato dall’auxina, suggerendo che questi due ormoni interagiscono per controllare lo sviluppo delle piante., In questa recensione, descriveremo la trasduzione del segnale di auxina e BR in base ai recenti progressi e esamineremo la diafonia tra auxina e crescita e sviluppo delle piante mediate da BR.
Via di segnalazione auxina
L’auxina è stata riconosciuta per la prima volta come ormone vegetale a causa del suo ruolo nel tropismo vegetale alla gravità o agli stimoli luminosi., Successivamente l’auxina è stata identificata chimicamente come acido indolo-3-acetico e ha dimostrato di svolgere ruoli essenziali in pletora di processi fisiologici e di sviluppo delle piante, tra cui embriogenesi, organogenesi, differenziazione vascolare, sviluppo di radici e germogli, crescita tropica e sviluppo della frutta (Estelle, 2011).
Utilizzando l’analisi genetica in Arabidopsis, il meccanismo molecolare alla base della trasduzione del segnale auxina è stato ben studiato. La RISPOSTA DELL’INIBITORE DEL TRASPORTO1 (TIR1) è stato il primo recettore nucleare identificato dell’auxina (Ruegger et al., 1998; Dharmasiri et al., 2005)., TIR1 codifica una proteina nucleare appartenente alla proteina F-box come subunità del complesso proteico SCF E3 ubiquitina ligasi (Gray et al., 1999, 2002; Hellmann et al., 2003; Quint et al., 2005), Oltre a TIR1, ci sono tre proteine F-box aggiuntive, ovvero le proteine F BOX DI SEGNALAZIONE AUXINA (AFBs) che mostrano attività di legame con l’auxina e mediano la segnalazione auxina in Arabidopsis (Badescu e Napier, 2006). Il recettore TIR1 può interagire con un gruppo di proteine AUX/IAA (auxina/acido indolo-3-acetico) (Dharmasiri et al., 2003)., Le proteine AUX/IAA sono regolatori negativi della segnalazione auxina e ci sono 29 membri di AUX / IAA codificati nel genoma di Arabidopsis. Le proteine AUX/IAA potrebbero interagire con la classe di regolatori trascrizionali, i fattori di risposta auxina (ARF), per mediare le risposte trascrizionali all’auxina. Sotto alto livello di auxina, le proteine AUX/IAA interagiscono con TIR1 come corecettore dell’auxina e possono essere ubiquitinate dal complesso SCFtir1 e quindi essere degradate attraverso la via ubiquitina-proteasoma (Gray et al., 2001; Lanza et al., 2012)., Dopo la distruzione dei repressori AUX/IAA, i regolatori trascrizionali auxina ARF che includono le appartenenze 23 possono essere rilasciati dalla repressione AUX/IAA e quindi mediare le risposte auxina mediante l’attivazione o la repressione dei geni bersaglio (Guilfoyle e Hagen, 2007). I diversi set di proteine F-box e AUX / IAA o ARFs deducono la complessità durante la trasduzione del segnale auxina (Goh et al., 2012; Guilfoyle, 2015; Salehin et al., 2015).,
L’azione coordinata dei repressori trascrizionali Aux/IAA e dei fattori di trascrizione ARF produce complesse reti di regolazione genica che sono state riportate anche in Physcomitrella (Lavy et al., 2016). Recentemente, è stato scoperto che la subunità CULLIN1 (CUL1) della SCF interagisce con TIR1 e quindi regola la stabilità dei substrati TIR1 e la segnalazione auxina (Wang et al., 2016). L’interazione tra TIR1 e Aux/IAA è anche influenzata dalla conformazione spaziale di Aux/IaaS, controllata da una isomerasi ciclofila LRT2 nel riso (Jing et al., 2015)., IL FATTORE DI SHOCK TERMICO 90 (HSP90) e il co-chaperone SGT1, rispettivamente, interagiscono con TIR1 e quindi regola la stabilità TIR1, che influenza le interazioni tra TIR1 e Aux/IAA e la segnalazione auxina (Wang et al., 2016).
Oltre alla via di segnalazione auxina canonica dipendente da TIR1, è stato recentemente riportato che l’auxina suscita una vasta gamma di risposte di sviluppo attraverso un meccanismo di segnalazione auxina non canonico., In questo processo di rilevamento dell’auxina non canonico, ARF3 / ETTIN controlla l’espressione genica attraverso interazioni con fattori di trascrizione specifici del processo, che arricchiscono altamente la diversità dello sviluppo vegetale mediata dall’auxina (Simonini et al., 2016, 2017).
BR Signaling Pathway
Il BRASSINOSTEROIDE è stato scoperto per la prima volta nel polline per la sua capacità di promuovere l’allungamento cellulare. Più tardi si è scoperto che BR svolge un ruolo in una vasta gamma di aspetti di crescita delle piante e può rispondere agli stress biotici e abiotici., Al giorno d’oggi la via di trasduzione del segnale BR è stata ampiamente chiarita da combinazioni di metodi diversi, tra cui genetica molecolare, biochimica, proteomica e genomica, ecc. La chinasi della superficie cellulare BRASSINOSTEROID INSENSITIVE1 (BRI1) è stata identificata come il recettore di BR che può legarsi al dominio extracellulare di BRI1 e attivare la sua attività chinasica e quindi attivare una cascata di segnale per regolare la trascrizione (Li e Chory, 1997; Wang et al., 2001; Kinoshita et al., 2005; Kim e Wang, 2010; Clouse, 2011; Hothorn et al., 2011; Lei et al., 2011; Oh et al., 2012)., Al momento della percezione di BR, BRI1 interagisce con la CHINASI 1 ASSOCIATA al co-recettore BRI1 (BAK1) e le sue EMBRIOGENESI SOMATICHE omolog CHINASi dei RECETTORI (SERKs) per formare un complesso recettoriale BR più attivo (Li et al., 2002; Nam e Li, 2002; Wang et al., 2005; Tang et al., 2008; Gou et al., 2012). BRI1 attivato fosforila due substrati di chinasi citoplasmatiche simili a recettori ancorati alla membrana plasmatica: CHINASI DI SEGNALAZIONE BRASSINOSTEROIDE1 (BSK1) e CRESCITA DIFFERENZIALE COSTITUTIVA1 (CDG1) (Tang et al., 2008; Kim et al.,, 2011), che a sua volta fosforila una fosfatasi di tipo PP1 denominata BRI1-SUPPRESSOR1 (BSU1) per attivare BSU1, portando alla defosforilazione di BSU1 e all’inattivazione della chinasi BRASSINOSTEROID INSENSITIVE2 (BIN2) simile a GSK3. L’attività della chinasi di BIN2 inoltre è inibita dalla DEACETILASI HDA6 dell’ISTONE, che interagisce e deacetylates al K189 di BIN2., Quando i livelli di BR sono bassi, BRI1 è quiescente a causa del suo regolatore negativo, dell’INIBITORE della CHINASI BRI1 1 (BKI1) e della fosfatasi proteica 2A (PP2A), mentre BIN2 fosforila due fattori di trascrizione omologhi BR, BRASSINAZOLO RESISTANT1 (BZR1) e BZR2 (chiamato anche BES1 per BRI1-EMS-SOPPRESSORE 1) (He et al., 2002; Wang et al., 2002; Yin et al., 2002; Mora-Garcia et al., 2004; Kim et al., 2009, 2011; Kim e Wang, 2010). Quando i livelli di BR sono alti, BIN2 è inattivato e BZR1 e BZR2 sono defosforilati da PP2A e si muovono nel nucleo per alterare l’espressione di migliaia di geni di risposta BR (He et al.,, 2005; Yin et al., 2005; Sun et al., 2010; Tang et al., 2011; Yu et al., 2011).
La sinergia tra segnalazione BR e Auxina
I percorsi di segnale auxina e BR svolgono ruoli diversi, tuttavia, hanno anche mostrato interazioni sinergiche e interdipendenti in una vasta gamma di processi di sviluppo. Ad esempio, sia i segnali auxina che BR possono promuovere l’espansione cellulare e possono interagire sinergicamente per promuovere l’allungamento degli ipocotili (Nemhauser et al., 2004)., La risposta di uno dei due percorsi nel promuovere l’allungamento dell’ipocotile richiede la funzione dell’altro e l’interdipendenza tra le vie BR e auxina (Nemhauser et al., 2004). L’auxina ha aumentato la lunghezza dell’ipocotile nelle piante di tipo selvaggio ma non nel mutante BR-insensibile bri1-116, e questo fenotipo insensibile all’auxina di bri1-116 è stato soppresso dal mutante dominante bzr1-1D, indicando che BR o BZR1 attivo è necessario per la promozione dell’auxina dell’allungamento ipocotilico., È stato trovato che la segnalazione di BR converge con il SOPPRESSORE DEL FITOCROMO B4-3 (SOB3) per controllare l’allungamento cellulare e la crescita ipocotilica attraverso la regolazione della PICCOLA AUXINA indotta dall’auxina FINO all’espressione RNA19 (SAUR19) (Favero et al., 2017). D’altra parte, il fattore di trascrizione regolato auxina SMALL ORGAN SIZE 1 (SMOS1) è stato recentemente trovato per controllare l’espansione cellulare attraverso l’interazione diretta con SMOS2/DLT, un membro della famiglia GRAS di co-regolatori trascrizionali che svolge un ruolo positivo nella segnalazione BR nel riso (Kim et al., 2009; Tong et al., 2012; Hirano et al., 2017)., I mutanti correlati all’auxina come iaa3 e arf6/arf8 erano meno sensibili al BR di quanto non fosse wild-type per l’allungamento dell’ipocotile e abolivano l’ipersensibilità di bzr1-1D all’auxina, suggerendo che la promozione di BR e BZR1 dell’allungamento dell’ipocotile richiede ARF6/8. L’analisi genome-wide ChIP-Seq ha rivelato che ARF6 condivide un vasto numero di target genomici (circa il 50%) con BZR1 e il fattore di trascrizione regolato dalla luce/temperatura PIF4 mediante analisi CHIP-Seq (Oh et al., 2014)., BZR1 e PIF4 interagiscono con ARF6 e attivano geni target condivisi legandosi a geni target condivisi cooperativamente durante l’allungamento degli ipocotili (Oh et al., 2014) e molti di questi geni bersaglio sovrapposti codificano le proteine della parete cellulare coinvolte nell’espansione cellulare.
Brassinosteroid e auxina svolgono anche ruoli importanti nel mantenimento del meristema apicale della radice (RAM) (Durbak et al., 2012). La RAM è costituita da un piccolo gruppo di cellule raramente divisorie noto come centro quiescente (QC), circondato da cellule staminali che danno origine ai vari tipi di tessuto toot., Il mantenimento della popolazione di cellule staminali della radice è regolato da HOMEOBOX 5 CORRELATO a WUSCHEL (WOX5) (Sarkar et al., 2007). WOX5 è limitato al controllo di qualità tramite segnalazione auxina e facilita la corretta espressione dei geni PLT (Aida et al., 2004; Ding e Friml, 2010). Le mutazioni nel gene del recettore BR BRASSINOSTEROID INSENSITIVE 1 (BRI1) provocano una progressione aberrante del ciclo cellulare nella RAM e causano una RAM più piccola (Gonzalez-Garcia et al., 2011; Hacham et al., 2011). L’auxina è nota per stimolare la biosintesi di BR (Chung et al.,, 2011), ma l’attività di BR non influenza l’espressione dei geni PIN (Hacham et al., 2011). I fenotipi di punta della radice dei mutanti BR non mostrano lo stesso dei mutanti auxina (Gonzalez-Garcia et al., 2011), indicando che il BR agisce sulla RAM indipendentemente dall’auxina.
I segnali brassinosteroid e auxina sono anche sinergicamente richiesti nella formazione di pattern radiali di fasci vascolari (Ibanes et al., 2009)., Dalle combinazioni di modellazione matematica e esperimenti biologici, auxina maxima, stabilita dal trasporto polare asimmetrico auxina, ma non cambiamenti sui livelli di auxina è importante per il posizionamento dei fasci vascolari. Segnale BR è stato dimostrato di servire come un segnale di promozione per il numero di cellule nell’anello provascolare che sono coerenti con auxina maxima. Quindi l’istituzione di una disposizione periodica dei fasci vascolari nelle riprese è sotto l’azione coordinata di questi due ormoni vegetali (Ibanes et al., 2009)., Entrambi i segnali sono anche coinvolti nello sviluppo della radice della pianta e l’interazione di BR e auxina è mediata da BREVIS RADIX (BRX) durante questo processo. BRX è importante per la biosintesi limitante di BR e l’applicazione esogena di BR può salvare i difetti mutanti di brx. Inoltre, l’espressione genica sensibile all’auxina è globalmente compromessa nel mutante brx e l’espressione di BRX è fortemente indotta dall’auxina e soppressa da BR, implicando che la biosintesi BR e la segnalazione auxina sono collegate attraverso un ciclo di feedback che coinvolge BRX durante lo sviluppo della radice (Mouchel et al., 2006).,
I brassinosteroidi e l’auxina svolgono anche ruoli sinergici durante lo sviluppo laterale della radice. Le BR funzionano principalmente all’inizio della radice primordiale laterale mentre l’auxina è necessaria sia per le fasi di iniziazione che di emergenza della formazione della radice laterale (Casimiro et al., 2001; Bhalerao et al., 2002; Benkova et al., 2003; Bao et al., 2004). Durante questi processi, BRS aumentare LRP iniziazione promuovendo acropetal trasporto auxina nella radice, ma non influenzando livello endogeno IAA (Bao et al., 2004)., Tutti questi rapporti suggeriscono che la diafonia tra BR e auxina svolge un ruolo importante nella crescita e nello sviluppo delle piante di regolazione.
BR Regola la segnalazione auxina
Oltre all’interdipendenza e alla cooperazione dei segnali auxina e BR durante lo sviluppo dell’impianto, BR potrebbe mediare la via del segnale auxina su più livelli. BZR1 interagisce con le proteine ARF per indirizzare direttamente più componenti di segnalazione auxina e geni coinvolti nel metabolismo auxina come il trasporto e la segnalazione, tra cui AUX/IAA, PIN, TIR1 e ARFs, ecc. (Sun et al., 2010)., È stato scoperto che le proteine Aux/IAA sono coinvolte nelle risposte BR e i mutanti iaa7/axr2-1 e iaa17/axr3-3 hanno mostrato una sensibilità BR aberrante e un’espressione genica aberrante indotta da BR in modo dipendente dall’organo (Nakamura et al., 2006). Il trattamento esogeno del brassinolide (BL) potrebbe indurre l’espressione dei geni auxina-rispondenti quali IAA5, IAA19, IAA17, ecc., e l’espressione dei geni di cui sopra è down-regolata nel mutante biosintetico BR de-etiolated2 (det2), che indica che la biosintesi funzionale BR è in parte richiesta per l’espressione genica dipendente dall’auxina (Nakamura et al.,, 2003; Kim et al., 2006). Inoltre, BR influisce anche sul flusso di auxina regolando l’espressione degli esportatori di auxina come PIN4 e PIN7 (Nakamura et al., 2004). Durante le risposte di gravitropismo vegetale, BRs potrebbe migliorare l’accumulo polare dell’esportatore di auxina PIN2 nella zona del meristema radicale e quindi influenzare la ridistribuzione dell’auxina dalla punta della radice verso le zone di allungamento e provocare la differenza dei livelli IAA in entrambi i lati superiore e inferiore delle radici per indurre il gravitropismo vegetale., Durante questo processo, BR attivato ROP2 svolge un ruolo importante nel modulare la localizzazione funzionale di PIN2 attraverso la regolazione del montaggio / rimontaggio di F-actins (Li et al., 2005). Ulteriori studi hanno dimostrato che una diminuzione della percezione e/o della concentrazione di BL potrebbe indurre il CYP79B2, il gene che codifica per un enzima che converte il triptofano in indolo-3-acetaldoxima e quindi influenzare la distribuzione (Kim et al., 2007).
Inoltre, è stato riscontrato che il segnale BR potrebbe regolare l’uscita di segnalazione auxina dal suo regolatore negativo GSK3 chinasi BIN2., Il fattore di risposta auxina ARF2 è stato identificato come una proteina interagente BIN2 in uno schermo a due ibridi di lievito e il test della chinasi ha mostrato che BIN2 poteva fosforilare ARF2. La fosforilazione di ARF2 provoca la perdita della sua capacità di legame del DNA e l’attività di repressione dei geni bersaglio (Vert et al., 2008). ARF2 è un gene bersaglio BZR1 e la sua espressione è ridotta dal trattamento BR (Sun et al., 2010)., Inoltre, BIN2 può fosforilare ARF7 e ARF19 per sopprimere la loro interazione con AUX / IaaS e quindi migliorare l’attività trascrizionale sui loro geni bersaglio CONFINI DEGLI ORGANI LATERALI-DOMAIN16 (LBD16) e LBD29 per regolare l’organogenesi laterale della radice (Cho et al., 2014). Tuttavia, BR svolge un ruolo minore durante questo processo e BIN2 è sotto il controllo del modulo INIBITORIO DEL FATTORE INIBITORIO DI DIFFERENZIAZIONE dell’ELEMENTO TRACHEALE (TDIF)–RECETTORE TDIF (TDR) (Cho et al., 2014). Insieme, BR può regolare le risposte auxina influenzando diversi componenti di segnalazione auxina.,
L’auxina regola la segnalazione BR
D’altra parte, l’auxina può anche regolare la via del segnale BR in determinati aspetti. L’espressione di DWARF4, un’idrossilasi cruciale per la biosintesi di BR per controllare il livello endogeno di BR, è auxina dipendente. Il trattamento con auxina potrebbe stimolare notevolmente l’espressione di DWARF4 e l’auxina potrebbe inibire il legame di BZR1 al promotore di DWARF4. L’induzione di DWARF4 da auxina richiede la via di segnalazione auxina ma non la via di segnalazione BR (Chung et al., 2011; Yoshimitsu et al., 2011)., CPD catalizzando l’ossidazione C-3 di BR è stata attivata da BRX, un fattore di trascrizione putativo che agisce a valle della segnalazione auxina (Mouchel et al., 2006). Ulteriori studi sul riso indicano che l’auxina esogena può migliorare i livelli di espressione della trascrizione del gene del recettore BR OsBRI1, suggerendo che l’auxina migliora la segnalazione BR attraverso la regolazione dei recettori BR (Sakamoto et al., 2013). Inoltre, il promotore di OsBRI1 possiede un motivo a monte dell’elemento auxina-risposta (AuxRE) che è preso di mira dai fattori di trascrizione ARF., Inoltre, gli studi sui mutanti indicano che dopo la mutazione di AuxRE, l’induzione dell’espressione di OsBRI1 da parte dell’auxina viene abolita e anche l’espressione di OsBRI1 è regolata in arf mutant (Sakamoto et al., 2013). È stato riportato che OsARF19 si lega al promotore di OsBRI1 e regola positivamente la sua espressione che quindi attiva la segnalazione BR (Zhang et al., 2015). BES1 può legarsi al promotore del PICCOLO RNA AUXINA-UP 15 (SAUR15) e mediare il gene di risposta precoce BR in Arabidopsis, e questo legame potrebbe essere migliorato dal trattamento con auxina (Walcher e Nemhauser, 2012)., Presi insieme, l’auxina può anche influenzare le risposte BR e la crescita e lo sviluppo delle piante regolate BR.
Osservazioni conclusive e prospettive future
Nel corso degli ultimi quasi quattro decenni, gli studi sulle interazioni pathway auxina-BR hanno attirato sempre più l’interesse dei ricercatori. L’apparecchio di strumenti fisiologici, molecolari, genetici e biochimici ha notevolmente approfondito la nostra comprensione di questo problema., Sulla base degli studi precedenti, BR e auxina sono coinvolte sinergicamente in molteplici processi di sviluppo vegetale, tra cui: allungamento ipocotilico, sviluppo di fasci vascolari, sviluppo di radici e tropismi, ecc. L’interdipendenza e la cooperazione di auxina e BR sono complicate e coinvolgono numerosi processi a livello molecolare, condividendo gli stessi geni bersaglio, regolandosi reciprocamente su più livelli (Figura 1).
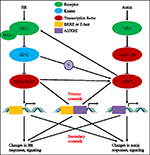
FIGURA 1. Modello di auxina-brassinosteroid (BR) crosstalk., Nell’Arabidopsis, le percezioni del segnale BR e auxina sono riconosciute rispettivamente dai recettori BRI1 e TIR1. BR si lega al dominio extracellulare di BRI1 e promuove interagisce con il co-recettore BAK1 per formare un complesso recettore BR più attivo, che a sua volta porta alla defosforilazione e all’inattivazione di BIN2. L’inattivazione di BIN2 porta alla defosforilazione di due fattori di trascrizione omologhi BR BZR1 e BZR2, che si muovono nel nucleo per attivare la trascrizione di geni contenenti BRRE o E-box nella loro regione promotrice., BIN2 può anche fosforilare ARF7 e ARF19 per sopprimere la loro interazione con AUX / IaaS e quindi migliorare l’attività trascrizionale sui loro geni bersaglio. TIR1 riceve la segnalazione auxina e interagisce con le proteine AUX/IAA come co-recettore dell’auxina. L’AUX / IAA viene quindi degradato attraverso la via ubiquitina–proteasoma e i regolatori trascrizionali dell’auxina i fattori di risposta all’auxina (ARFs) vengono rilasciati dalla repressione AUX/IAA e attivano la trascrizione dei geni con elementi reattivi all’auxina (AUXRE) nella loro regione regolatrice., Alcuni ARFS possono anche legarsi al promotore di BRI1 e regola positivamente la sua espressione che poi attiva la segnalazione di BR. La diafonia primaria si verifica mediante l’attivazione di geni che contengono sia BRRE/E-box che AUXRE nella loro regione promotrice, consentendo a entrambe le vie di segnalazione di regolare direttamente la trascrizione. La diafonia secondaria avviene attraverso l’espressione di geni che sono auxina o BR reattivi, ma le cui attività controllano l’espressione di geni che regolano la risposta e la segnalazione di altri ormoni.,
La regolazione della fosforilazione svolge un ruolo cruciale nella via di segnalazione BR, specialmente durante il processo di percezione, BR viene percepito attraverso il recettore della chinasi BRI1 e i co-recettori della chinasi BAK1, e alla fine controlla l’espressione genica regolata da BR attraverso l’influenza di fattori di trascrizione a valle, 2005; Yin et al., 2005; Sun et al., 2010; Tang et al., 2011; Yu et al., 2011). Tuttavia, la regolazione dell’ubiquitinazione sembra essenziale per la segnalazione auxina., Una volta che l’auxina si lega al recettore TIR1, che agisce come un’ubiquitina E3-ligasi, la TIR1 E3-ligasi attivata ubiquita le proteine AUX/IAA, porta alla degradazione di questi repressori e de-reprime i fattori di trascrizione ARF e alla fine causa cambiamenti del pattern di espressione genica regolati dall’auxina e risposte alla crescita (Gray et al., 1999, 2002; Hellmann et al., 2003; Quint et al., 2005). Poiché è stato trovato che la chinasi BIN2, che è ben nota funzionare nella segnalazione BR, potrebbe fosforilare e migliorare le attività di ARF come ARF2 e ARF7 (Vert et al., 2008; Cho et al.,, 2014), sarà interessante testare se le chinasi come BIN2, che sono coinvolte nella segnalazione BR, potrebbero anche interagire con altri componenti di segnalazione auxina come il recettore TIR1 o i repressori AUX/IAA e influenzare l’attività della TIR1 E3-ligasi o le stabilità proteiche AUX/IAA. D’altra parte, anche il ruolo dell’ubiquitinazione nella segnalazione BR deve essere affrontato, specialmente se TIR1 E3-ligasi potrebbe interagire direttamente con i componenti di segnalazione BR e regolare le loro stabilità proteiche.,
Inoltre, utilizzando la risposta auxina DR5 e altri reporter auxina, è stato osservato che l’auxina regola la crescita e lo sviluppo delle piante in modo tissutale o cellulare dipendente. Le diverse uscite trascrizionali a seconda del contesto cellulare e ambientale (Clark et al., 2014; Etchells et al., 2016; Lavy et al., 2016)., Sebbene la segnalazione spaziotemporale BR abbia dimostrato di controllare la crescita delle radici attraverso l’azione antagonista con auxina (Chaiwanon e Wang, 2015), non è ancora noto se la segnalazione tissutale o cellulare BR, che potrebbe essere visualizzata da pBZR1:BZR1-YFP, è anche importante per controllare altri processi oltre allo sviluppo delle radici. Inoltre, la generazione di una mappa dettagliata del tessuto o della cantina delle distribuzioni di auxina e BR è attualmente possibile utilizzando lo smistamento cellulare attivato dalla fluorescenza o la microdissezione laser in combinazione con l’analisi dell’espressione genica ad alta risoluzione., Questo alla fine porta ad affrontare se il crosstalks auxina con BR in un tessuto o modo cellulare.
Contributi dell’autore
Tutti gli autori sono stati coinvolti nella stesura di questo manoscritto di recensione. Per ulteriori informazioni su ciò che costituisce la paternità, si prega di fare riferimento alle nostre linee guida autore.,
Finanziamento
Questo lavoro è supportato da sovvenzioni della National Natural Science Foundation of China (Progetti 31500229 e 31770305), Shandong Province Natural Science Foundation of Major Basic Research Program (2017C03), Shandong Provincial Funds for Distinguished Young Scholars (2014JQ201408).
Dichiarazione sul conflitto di interessi
Gli autori dichiarano che la ricerca è stata condotta in assenza di relazioni commerciali o finanziarie che potrebbero essere interpretate come un potenziale conflitto di interessi.
Clouse, S. D. (2011)., Trasduzione del segnale brassinosteroide: dall’attivazione della chinasi del recettore alle reti trascrizionali che regolano lo sviluppo delle piante. Cellula vegetale 23, 1219-1230. doi: 10.1105 / tpc.111.084475
PubMed Abstract | CrossRef Full Text/Google Scholar
Estelle, M. (2011). Segnalazione auxina: dalla sintesi alla biologia dei sistemi; Una raccolta di soggetti da Cold Spring Harbor Perspectives in Biology. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press.
Google Scholar
Guilfoyle, TJ (2015)., The PB1 domain in auxin response factor and Aux/IAA proteins: a versatile protein interaction module in the auxin response. Plant Cell 27, 33–43. doi: 10.1105/tpc.114.132753
PubMed Abstract | CrossRef Full Text | Google Scholar