wprowadzenie
w przeciwieństwie do zwierząt, które mogą poruszać się, aby uniknąć niekorzystnego otoczenia, rośliny siedzące wykazują wysoce rozwiniętą adaptację do skomplikowanych warunków środowiskowych. Aby osiągnąć tę głęboką zdolność adaptacyjną, niezbędna jest komunikacja między komórkami. Komunikacja komórkowa w roślinach obejmuje solidne wewnątrzkomórkowe przetwarzanie sygnałowe i skomplikowane międzykomórkowe sieci sygnałowe., Do tej pory istnieje co najmniej dziewięć substancji sygnalizacyjnych, zwanych hormonami roślinnymi, w tym auksyna, brassinosteroid (BR), cytokinina, gibereliny (GA), etylen, kwas jasmonowy (JA), strigolakton (SL), kwas abscysynowy (ABA) i kwas salicylowy (SA) odkryte (Druege et al., 2016; Verma et al., 2016). Badania genetyczne i fizjologiczne ujawniły krytyczne role i mechanizmy funkcjonalne tych hormonów w wzroście i rozwoju roślin (Gray, 2004)., Na podstawie wcześniejszych badań auksyna, BR, GA, SL i cytokinina działają głównie podczas normalnego wzrostu i rozwoju roślin, podczas gdy ABA, etylen, ja i SA odgrywają ważną rolę w odpowiedzi wzrostu roślin na różne naprężenia biotyczne i abiotyczne (Pieterse et al., 2009; Santner i in., 2009; Denance et al., 2013). A także niektóre z tych hormonów mają podwójną rolę, na przykład ABA odgrywa również ważną rolę w rozwoju nasion i uśpieniu (Seo and Koshiba, 2002)., Chociaż każdy hormon odgrywa dominującą rolę w pewnych aspektach, wiele hormonów nakładało się na działania i interakcje różnych hormonów kontrolują wiele aspektów rozwojowych i wzrostu w odpowiedzi na endogenne sygnały rozwojowe i egzogenne.
Auksyna i BR to dwie główne klasy hormonów stymulujących wzrost., BR, grupa hormonów steroidowych specyficznych dla roślin, które mogą wchodzić w interakcje z innymi fitohormonami, takimi jak auksyna, cytokinina, etylen, GA, JA i SA i regulują szeroki zakres procesów wzrostu i rozwoju roślin, w tym kiełkowanie nasion, wydłużanie komórek, różnicowanie naczyniowe, tworzenie i ruch szparek, kwitnienie i płodność męska (Saini et al., 2015). Co ciekawe, każdy z tych procesów jest również kontrolowany przez auksyny, co sugeruje, że te dwa hormony współdziałają w celu kontrolowania rozwoju roślin., W tym przeglądzie przedstawimy transdukcję sygnału auksyny I BR na podstawie ostatnich postępów i przeglądu przesłuchu między auksyną a br pośredniczy wzrost i rozwój roślin.
szlak sygnałowy auksyny
Auksyna została po raz pierwszy uznana za hormon roślinny ze względu na jego rolę w tropizmie roślin do bodźców grawitacyjnych lub świetlnych., Później auksyna została chemicznie zidentyfikowana jako kwas indol-3-octowy i wykazano, że odgrywa istotną rolę w wielu procesach rozwojowych i fizjologicznych roślin, w tym embriogenezie, organogenezie, różnicowaniu naczyniowym, rozwoju korzeni i pędów, wzroście tropicznym i rozwoju owoców (Estelle, 2011).
wykorzystując analizę genetyczną Arabidopsis, dobrze zbadano mechanizm molekularny leżący u podstaw transdukcji sygnału auksyny. Odpowiedź inhibitora TRANSPORTU1 (TIR1) był pierwszym zidentyfikowanym receptorem jądrowym auksyny (Ruegger et al., 1998; Dharmasiri et al., 2005)., TIR1 koduje białko jądrowe należące do białka F-box jako podjednostkę kompleksu białkowego ligazy ubikwityny SCF E3 (Gray et al., 1999, 2002; Hellmann et al., 2003; Quint et al., 2005), oprócz TIR1, istnieją trzy dodatkowe białka F-box, mianowicie AUXIN SIGNALING F Box PROTEINs (AFBs), które wykazują aktywność wiązania auksyny i pośredniczą w sygnalizacji auksyny w Arabidopsis (Badescu i Napier, 2006). Receptor TIR1 może wchodzić w interakcje z grupą białek AUX/IAA (auksyna/kwas indol-3-octowy) (Dharmasiri et al., 2003)., Białka AUX / IAA są negatywnymi regulatorami sygnalizacji auksyny, a w genomie Arabidopsis znajduje się 29 członków AUX/IAA. Białka AUX / IAA mogą wchodzić w interakcje z klasą regulatorów transkrypcyjnych, współczynników odpowiedzi auksyny (ARF), aby pośredniczyć w odpowiedzi transkrypcyjnej na auksynę. Pod wysokim poziomem auksyny białka AUX/IAA oddziałują z TIR1 jako coreceptor auksyny i mogą być ubikwitynowane przez kompleks SCFtir1, a tym samym ulegać degradacji poprzez szlak ubikwityny-proteasomu (Gray et al., 2001; Lanza et al., 2012)., Po zniszczeniu represji AUX/IAA z represji AUX/IAA mogą zostać uwolnione regulatory transkrypcji aux, które obejmują 23 członków, a tym samym pośredniczą w odpowiedzi auksyny poprzez aktywację lub represję genów docelowych (Guilfoyle and Hagen, 2007). Różne zestawy białka F-box i AUX / IAA lub ARFs wnioskują złożoność podczas transdukcji sygnału auxin (Goh et al., 2012; Guilfoyle, 2015; Salehin et al., 2015).,
skoordynowane działanie represji transkrypcyjnych Aux / IAA i czynników transkrypcyjnych ARF wytwarza złożone sieci regulacji genów, które również opisano w Physcomitrella (Lavy et al., 2016). Niedawno stwierdzono, że podjednostka CULLIN1 (CUL1) SCF oddziałuje z TIR1, a tym samym reguluje stabilność substratów TIR1 i sygnalizację auksyny (Wang et al., 2016). Interakcja między TIR1 i Aux / IAA jest również pod wpływem przestrzennej konformacji Aux / IAAs, kontrolowanej przez izomerazę cyklofilinową LRT2 w ryżu (Jing et al., 2015)., Współczynnik szoku cieplnego 90 (HSP90) i współodczuwacz SGT1, odpowiednio, współdziała z TIR1, a tym samym reguluje stabilność TIR1, co wpływa na interakcje między TIR1 i AUX / IAA i sygnalizacją auksyny (Wang et al., 2016).
oprócz zależnego od TIR1 kanonicznego szlaku sygnałowego auksyny, niedawno donoszono, że auksyna wywołuje zróżnicowany zakres odpowiedzi rozwojowych poprzez niekanoniczny mechanizm sygnałowy auksyny., W tym niekanonicznym procesie wykrywania auksyny, arf3 / ETTIN kontroluje ekspresję genów poprzez interakcje z specyficznymi dla procesu czynnikami transkrypcyjnymi, co bardzo wzbogaca różnorodność rozwojową roślin za pośrednictwem auksyny (Simonini et al., 2016, 2017).
Br>
BRASSINOSTEROID został po raz pierwszy odkryty w pyłku ze względu na jego zdolność do promowania wydłużenia komórek. Później stwierdzono, że BR odgrywa rolę w szerokim zakresie aspektów wzrostu roślin i może reagować na naprężenia biotyczne i abiotyczne., Obecnie szlak transdukcji sygnału BR został w dużej mierze wyjaśniony za pomocą kombinacji różnych metod, w tym genetyki molekularnej, biochemii, proteomiki i genomiki itp. Niewrażliwość na kinazę na powierzchni komórki (BRI1) została zidentyfikowana jako receptor BR, który może wiązać się z pozakomórkową domeną BRI1 i aktywować jej aktywność kinazy, a tym samym włączać kaskadę sygnałową w celu regulacji transkrypcji (Li and Chory, 1997; Wang et al., 2001; Kinoshita et al., 2005; Kim i Wang, 2010; Clouse, 2011; Hothorn et al., 2011; ona i inni., 2011; Oh et al., 2012)., Po percepcji BR, BRI1 wchodzi w interakcję z kinazą 1 skojarzoną z receptorem BRI1 (BAK1) i jej kinazami HOMOLOGICZNYMI EMBRIOGENESISRECEPTOR (SERKs), tworząc bardziej aktywny kompleks receptora BR (Li et al., 2002; Nam and Li, 2002; Wang et al., 2005; Tang et al., 2008; Gou i in., 2012). Aktywowany bri1 fosforyluje dwa substraty kinaz cytoplazmatycznych zakotwiczonych w błonie osocza receptorów: kinazy sygnałowe BRASSINOSTEROIDA1 (BSK1) i konstytutywny wzrost RÓŻNICOWY1 (CDG1) (Tang et al., 2008; Kim i in.,, 2011), który z kolei fosforyluje fosfatazę typu PP1 o nazwie BRI1-SUPRESOR1 (BSU1), aby aktywować BSU1, prowadząc do defosforylacji bsu1 i inaktywacji niewrażliwości na kinazę BRASINOSTEROIDOWĄ GSK32 (BIN2). Aktywność kinazy BIN2 jest również hamowana przez DEACETYLAZĘ HISTONOWĄ HDA6, która oddziałuje i deacetyluje w K189 BIN2., Gdy poziomy BR są niskie, BRI1 jest spokojne ze względu na jego ujemny regulator, INHIBITOR kinazy BRI1 1 (BKI1) i fosfatazy białkowej 2A (PP2A), podczas gdy BIN2 fosforyluje dwa homologiczne czynniki transkrypcyjne br, BRASSINAZOLE RESISTANT1 (BZR1) i BZR2 (zwany również BES1 dla BRI1-EMS-supresor 1) (He et al., 2002; Wang et al., 2002; Yin et al., 2002; Mora-Garcia et al., 2004; Kim i in., 2009, 2011; Kim i Wang, 2010). Gdy poziomy BR są wysokie, BIN2 jest inaktywowany, i BZR1 i BZR2 są defosforylowane przez PP2A, i przenieść do jądra, aby zmienić ekspresję tysięcy genów odpowiedzi BR (He et al.,, 2005; Yin et al., 2005; Sun et al., 2010; Tang et al., 2011; Yu i in., 2011).
synergia pomiędzy sygnałem Br i Auksyną
ścieżki sygnałowe auksyny i BR odgrywają różną rolę, jednak wykazały również synergistyczne i współzależne interakcje w szerokim zakresie procesów rozwojowych. Na przykład, zarówno auxin i BR sygnały mogą promować ekspansję komórek i mogą oddziaływać synergistycznie w celu promowania wydłużenia hipokotyli (Nemhauser et al., 2004)., Reakcja jednego z dwóch szlaków w promowaniu elongacji hipokotylowej wymaga funkcji drugiego i współzależności między szlakami Br i auksyny (Nemhauser et al., 2004). Auksyna zwiększała długość hipokotylu u roślin dziko żyjących, ale nie u niewrażliwego na BR mutanta bri1-116, A fenotyp niewrażliwego na BR1-116 został stłumiony przez dominujący Mutant bzr1-1D, co wskazuje, że BR lub aktywny BZR1 jest wymagany do promowania wydłużenia hipokotylu przez auksyny., Stwierdzono, że sygnalizacja BR zbiega się z tłumikiem FITOCHROMU B4-3 (SOB3) w celu kontrolowania wydłużenia komórek i wzrostu hipokotylu poprzez regulację indukowanej auksyny małej ekspresji auksyny RNA19 (SAUR19) (Favero et al., 2017). Z drugiej strony, czynnik transkrypcyjny regulowany przez auksyny small ORGAN SIZE 1 (SMOS1) niedawno stwierdzono, aby kontrolować ekspansję komórek poprzez bezpośrednią interakcję z SMOS2/DLT, członkiem rodziny Gras transkrypcyjnych współregulatorów, który odgrywa pozytywną rolę w sygnalizacji BR w ryżu (Kim et al., 2009; Tong et al., 2012; Hirano et al., 2017)., Mutanty związane z auksyną, takie jak iaa3 i arf6/arf8, były mniej wrażliwe na BR niż w przypadku elongacji hipokotylu typu dzikiego, i zniosły nadwrażliwość bzr1-1D na auksynę, co sugeruje, że promocja BR I BZR1 elongacji hipokotylu wymaga ARF6/8. Genomu ChIP-SEQ analiza ujawniła, że arf6 akcji ogromną liczbę celów genomicznych (około 50%) Z BZR1 i światło / regulowany czynnik transkrypcyjny PIF4 przez chip-SEQ analizy (Oh et al., 2014)., BZR1 i PIF4 oddziałują z ARF6 i aktywują wspólne geny docelowe poprzez wiązanie się ze współdzielonymi genami docelowymi współpracująco podczas elongacji hipokotyli (Oh et al., 2014) i wiele z tych nakładających się genów docelowych koduje białka ściany komórkowej zaangażowane w ekspansję komórkową.
Brassinosteroid i auksyna odgrywają również ważną rolę w utrzymaniu korzeniowego merystemu wierzchołkowego (RAM) (Durbak et al., 2012). Baran składa się z niewielkiej grupy rzadko dzielących się komórek znanych jako centrum spoczynkowe (QC), otoczonych komórkami macierzystymi, które powodują powstawanie różnych typów tkanek toot., Utrzymanie populacji korzeniowych komórek macierzystych jest regulowane przez WUSCHEL-RELATED HOMEOBOX 5 (WOX5) (Sarkar et al., 2007). WOX5 jest ograniczony do QC przez sygnalizację auksyny i ułatwia prawidłową ekspresję genów PLT (Aida et al., 2004; Ding and Friml, 2010). Mutacje w genie receptora BR BRASSINOSTEROID niewrażliwy 1 (BRI1) spowodować aberrant cyklu komórkowego progresji w barana i spowodować mniejsze Barana (Gonzalez-Garcia et al., 2011; Hacham et al., 2011). Wiadomo, że auksyna stymuluje biosyntezę BR (Chung et al.,, 2011), ale aktywność BR nie wpływa na ekspresję genów PIN (Hacham et al., 2011). Fenotypy końcówki korzenia mutantów BR nie wykazują tego samego, co mutanty auksyny (Gonzalez-Garcia et al., 2011), wskazując, że BR działa na baran niezależnie od auksyny.
sygnały Brassinosteroidu i auksyny są również synergistycznie wymagane w tworzeniu promienistego wzoru wiązek naczyniowych (Ibanes et al., 2009)., Dzięki połączeniu Modelowania Matematycznego i eksperymentów biologicznych, maksyma auksyny, ustalona przez asymetryczny transport auksyny polarnej, ale nie zmiany na poziomach auksyny jest ważna dla pozycjonowania wiązek naczyniowych. Wykazano, że sygnał BR służy jako sygnał promujący liczbę komórek w pierścieniu proweniencyjnym, które są zgodne z maksymą auksyny. Tak więc ustanowienie okresowego układu wiązek naczyniowych w pędu jest pod skoordynowanym działaniem tych dwóch hormonów roślinnych (Ibanes et al., 2009)., Oba sygnały są również zaangażowane w rozwój korzeni roślin, a interakcja BR i auksyny jest pośredniczona przez BREVIS RADIX (BRX) podczas tego procesu. BRX jest ważny dla ograniczającego szybkość biosyntezy BR I Br egzogenne zastosowanie może ratować brx Mutant defekty. Ponadto ekspresja genu reagującego na auksynę jest globalnie upośledzona u mutanta brx, a ekspresja BRX jest silnie indukowana przez auksynę i tłumiona przez BR, co sugeruje, że biosynteza BR i sygnalizacja auksynowa są połączone za pomocą pętli sprzężenia zwrotnego z BRX podczas rozwoju korzeni (Mouchel et al., 2006).,
Brassinosteroidy i auksyny odgrywają również rolę synergiczną podczas bocznego rozwoju korzeni. BRs działają głównie w inicjacji pierwotnej korzenia bocznego, podczas gdy auksyna jest wymagana zarówno w początkowej, jak i wschodzącej fazie tworzenia się korzenia bocznego (Casimiro et al., 2001; Bhalerao et al., 2002; Benkova et al., 2003; Bao et al., 2004). Podczas tych procesów BRs zwiększają inicjację LRP poprzez promowanie transportu akropetalu auksyny w korzeniu, ale nie poprzez wpływanie na poziom endogennego IAA (Bao et al., 2004)., Wszystkie te raporty sugerują, że przesłuchy między br a auksyną odgrywają ważną rolę w regulacji wzrostu i rozwoju roślin.
BR reguluje sygnalizację auksyny
oprócz współzależności i współpracy sygnałów auksyny I Br podczas rozwoju roślin, BR może pośredniczyć w szlaku sygnału auksyny na wielu poziomach. BZR1 oddziałuje z białkami ARF bezpośrednio na wiele elementów sygnałowych auksyny i genów zaangażowanych w metabolizm auksyny, takich jak transport i sygnalizacja, w tym AUX / IAA, piny, TIR1 i ARFs, itp. (Sun et al., 2010)., Stwierdzono, że białka Aux/IAA są zaangażowane w odpowiedzi BR, a mutanty iaa7/axr2-1 i iaa17/axr3-3 wykazywały nieprawidłową wrażliwość BR i nieprawidłową ekspresję genów indukowanych przez BR w sposób zależny od narządów (Nakamura et al., 2006). Egzogenne leczenie brasynolidem (BL) może wywoływać ekspresję genów reagujących na auksyny, takich jak IAA5, IAA19, IAA17 itp., a ekspresja powyższych genów jest regulowana w dół w biosyntetycznym mutancie br de-etiolated2 (det2), co wskazuje, że funkcjonalna biosynteza BR jest częściowo wymagana dla ekspresji genów zależnych od auksyny (Nakamura et al.,, 2003; Kim i in., 2006). Dodatkowo BR wpływa również na przepływ auksyny poprzez regulację ekspresji auksyny, takiej jak PIN4 i PIN7 (Nakamura et al., 2004). Podczas reakcji gravitropizmu roślinnego, BRs może zwiększyć polarną akumulację eksportera auksyny PIN2 w strefie merystemu korzenia, a tym samym wpłynąć na redystrybucję auksyny z końcówki korzenia w kierunku stref wydłużenia i spowodować różnicę poziomów IAA zarówno w górnej, jak i dolnej stronie korzeni, aby wywołać gravitropizm roślin., Podczas tego procesu, aktywowany przez br ROP2 odgrywa ważną rolę w modulowaniu funkcjonalnej lokalizacji PIN2 poprzez regulację montażu / ponownego montażu F-actyn (Li et al., 2005). Dalsze badania wykazały, że zmniejszona percepcja BL i / lub stężenie może indukować CYP79B2, gen kodujący enzym konwertujący tryptofan do indolu-3-acetaldoksym, a tym samym wpływać na dystrybucję (Kim et al., 2007).
Ponadto stwierdzono, że sygnał br może regulować wyjście sygnalizacji auksyny za pomocą ujemnego regulatora kinazy GSK3 BIN2., Czynnik reakcji auksyny ARF2 został zidentyfikowany jako białko oddziałujące na BIN2 w dwu-hybrydowym ekranie drożdży i teście kinaz wykazały, że BIN2 może fosforylować ARF2. Fosforylacja ARF2 powoduje utratę zdolności wiązania DNA i aktywność represji genów docelowych (Vert et al., 2008). ARF2 jest genem docelowym BZR1, a jego ekspresja jest zmniejszona przez Leczenie BR (Sun et al., 2010)., Dodatkowo BIN2 może fosforylować ARF7 i ARF19 w celu stłumienia ich interakcji z AUX/IAAs, a tym samym zwiększenia aktywności transkrypcyjnej na ich docelowych genach bocznych granic narządów-DOMAIN16 (LBD16) i LBD29 w celu regulacji bocznej organogenezy korzeni (Cho et al., 2014). Jednak BR odgrywa niewielką rolę podczas tego procesu, a BIN2 jest pod kontrolą czynnika hamowania różnicowania elementów TCHAWICZYCH–TDIF) – modułu receptora TDIF (TDR) (Cho et al., 2014). Razem BR może regulować reakcje auksyn poprzez wpływanie na różne komponenty sygnalizacyjne auksyny.,
Auxin reguluje sygnalizację BR
z drugiej strony auxin może również regulować ścieżkę sygnału BR w pewnych aspektach. Ekspresja DWARF4, kluczowej hydroksylazy dla biosyntezy BR do kontroli endogennego poziomu BR, jest zależna od auksyny. Leczenie auksyną może znacząco stymulować ekspresję karłowatości 4, A auksyna może hamować Wiązanie BZR1 z promotorem karłowatości 4. Indukcja DWARF4 przez auksynę wymaga szlaku sygnałowego auksyny, ale nie szlaku sygnałowego BR (Chung et al., 2011; Yoshimitsu i in., 2011)., CPD katalizujące utlenianie C-3 BR został aktywowany przez BRX, przypuszczalny czynnik transkrypcyjny działający poniżej sygnalizacji auksyny (Mouchel et al., 2006). Dalsze badania na ryżu wskazują, że egzogenna auksyna może zwiększać poziom ekspresji transkrypcji genu receptora Br OsBRI1, co sugeruje, że auksyna wzmacnia sygnalizację BR poprzez regulację receptorów BR (Sakamoto et al., 2013). Ponadto promotor OsBRI1 posiada motyw auxin-response element (auxre), który jest kierowany przez czynniki transkrypcyjne ARF., Co więcej, badania nad mutacjami wskazują, że po mutacji AuxRE indukcja ekspresji OsBRI1 przez auksynę jest zniesiona, a także ekspresja OsBRI1 jest regulowana w mutacji arf (Sakamoto et al., 2013). Donoszono, że OsARF19 wiąże się z promotorem OsBRI1 i pozytywnie reguluje jego ekspresję, która następnie aktywuje sygnalizację BR (Zhang et al., 2015). BES1 może wiązać się z promotorem małego auksyny RNA 15 (SAUR15) i mediować Gen wczesnej odpowiedzi BR u Arabidopsis, a wiązanie to może być wzmocnione przez leczenie auksyną (Walcher and Nemhauser, 2012)., Razem wzięte auksyny mogą również wpływać BR odpowiedzi i BR reguluje wzrost i rozwój roślin.
Uwagi końcowe i perspektywa na przyszłość
w ciągu ostatnich prawie czterech dekad badania nad interakcjami szlaku auksyny-br przyciągają coraz więcej badaczy. Wyposażenie fizjologicznych, molekularnych, genetycznych i biochemicznych narzędzi znacznie pogłębiło nasze zrozumienie tego problemu., Na podstawie wcześniejszych badań, Br i auksyny są zaangażowane synergistycznie w wiele procesów rozwojowych roślin, w tym: wydłużenie hypokotylu, rozwój wiązek naczyniowych, rozwój korzeni i tropizmów itp. Współzależność i współpraca auksyny I Br są skomplikowane i obejmują liczne procesy na poziomie molekularnym, poprzez dzielenie tych samych genów docelowych, regulujących się wzajemnie na wielu poziomach (ryc. 1).
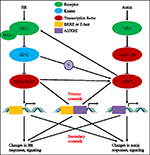
rysunek 1. Model Crosstalk auxin-brassinosteroid (BR)., W Arabidopsis percepcja sygnału Br i auksyny jest rozpoznawana odpowiednio przez receptory BRI1 i TIR1. BR wiąże się z pozakomórkową domeną BRI1 i promuje ją w interakcję z ko-receptorem BAK1, tworząc bardziej aktywny kompleks receptora BR, co z kolei prowadzi do defosforylacji i inaktywacji BIN2. Inaktywacja BIN2 prowadzi do defosforylacji dwóch homologicznych czynników transkrypcyjnych br BZR1 i BZR2, które przenoszą się do jądra komórkowego w celu aktywacji transkrypcji genów zawierających BRRE lub e-box w ich regionie promotora., BIN2 może również fosforylować ARF7 i ARF19 w celu zahamowania ich interakcji z AUX/IAAs, a tym samym zwiększenia aktywności transkrypcyjnej na ich genach docelowych. TIR1 odbiera sygnalizację auksyny I wchodzi w interakcje z białkami AUX/IAA jako Ko-receptor auksyny. AUX / IAA ulega degradacji poprzez szlak ubikwityny–proteasomu, a z represji AUX/IAA uwalniane są regulatory transkrypcji auxin response factors (arfs), które aktywują transkrypcję genów z elementami reagującymi na auxin (AUXRE) w ich regionie regulacyjnym., Niektóre ARFs mogą również wiązać się z promotorem BRI1 i pozytywnie regulować jego ekspresję, która następnie aktywuje sygnalizację BR. Pierwotne przesłuchanie zachodzi poprzez aktywację genów, które zawierają zarówno BRRE / e-box, jak i AUXRE w swoim regionie promotora, pozwalając obu szlakom sygnałowym bezpośrednio regulować transkrypcję. Wtórny crosstalk zachodzi poprzez ekspresję genów, które reagują na auksyny lub BR, ale których działania kontrolują ekspresję genów, które regulują odpowiedź i sygnalizację innych hormonów.,
Regulacja fosforylacji odgrywa kluczową rolę w szlaku sygnałowym BR, szczególnie podczas procesu percepcji, BR jest postrzegany przez receptor kinazy BRI1 i Ko-receptory kinazy BAK1, a ostatecznie kontroluje ekspresję genu regulowanego BR poprzez wpływanie na późniejsze czynniki transkrypcyjne, takie jak aktywność BES1 / BZR1 (He et al., 2005; Yin et al., 2005; Sun et al., 2010; Tang et al., 2011; Yu i in., 2011). Jednak regulacja ubikwitynacji wydaje się niezbędna dla sygnalizacji auksyny., Gdy auksyna wiąże się z receptorem TIR1, który działa jako ligaza ubikwityny E3, aktywowane tir1 E3-ligaza ubikwityny AUX / IAA białka, prowadzi do degradacji tych represji i de-represji ARF czynników transkrypcyjnych, i ostatecznie powoduje auksyny regulowane zmiany wzoru ekspresji genów i odpowiedzi wzrostu (Gray et al., 1999, 2002; Hellmann et al., 2003; Quint et al., 2005). Ponieważ stwierdzono, że kinaza BIN2, która jest dobrze znana funkcjonująca w sygnalizacji BR, może fosforylować i zwiększać aktywność ARF, takich jak ARF2 i ARF7 (Vert et al., 2008; Cho i in.,, 2014), interesujące będzie sprawdzenie, czy kinazy, takie jak BIN2, które są zaangażowane w sygnalizację BR, mogą również wchodzić w interakcje z innymi składnikami sygnalizacji auksyny, takimi jak receptor TIR1 lub represory AUX/IAA, I wpływać na aktywność ligazy TIR1 E3 lub stabilność białka AUX/IAA. Z drugiej strony należy zająć się rolą ubikwitynacji w sygnalizacji BR, zwłaszcza jeśli LIGAZA TIR1 E3 może bezpośrednio oddziaływać ze składnikami sygnalizacji BR i regulować ich stabilność białek.,
ponadto, przy użyciu reakcji auksyny DR5 i innych reporterów auksyny, zaobserwowano, że auksyna reguluje wzrost i rozwój roślin w sposób zależny od tkanki lub komórki. Różnorodne wyjścia transkrypcyjne w zależności od kontekstu komórkowego i środowiskowego (Clark et al., 2014; Etchells et al., 2016; Lavy et al., 2016)., Chociaż wykazano, że spatiotemporal br kontroluje wzrost korzeni poprzez antagonistyczne działanie auksyny( Chaiwanon and Wang, 2015), nadal nie wiadomo, czy sygnalizacja tkankowa lub Komórkowa BR, która może być wizualizowana przez pBZR1:BZR1-YFP, jest również ważna do kontrolowania innych procesów poza rozwojem korzeni. Ponadto, generowanie szczegółową tkankową lub Piwniczną mapę dystrybucji auksyny i BR jest obecnie możliwe przy użyciu fluorescencyjnego aktywowanego sortowania komórek lub laserowej mikrodissekcji w połączeniu z analizą ekspresji genów o wysokiej rozdzielczości., To ostatecznie prowadzi do adres jeśli auksyny crosstalks z BR w sposób tkankowy lub komórkowy.
wkład autora
wszyscy autorzy byli zaangażowani w pisanie tego recenzowanego manuskryptu. Aby uzyskać więcej informacji na temat tego, co stanowi autorstwo, zapoznaj się z naszymi wytycznymi dla autorów.,
finansowanie
ta praca jest wspierana przez granty z National Natural Science Foundation Of China (projekty 31500229 i 31770305), Shandong Province Natural Science Foundation głównego programu badań podstawowych (2017c03), Shandong Provincial Funds for Distinguished Young Scholars (2014JQ201408).
Oświadczenie o konflikcie interesów
autorzy oświadczają, że badanie zostało przeprowadzone w przypadku braku jakichkolwiek relacji handlowych lub finansowych, które mogłyby być interpretowane jako potencjalny konflikt interesów.
, Brassinosteroid transdukcja sygnału: od aktywacji kinazy receptorowej do sieci transkrypcyjnych regulujących rozwój roślin. Cela Roślinna 23, 1219-1230. podoba mi się! do obserwowanych nr: 101105111.084475
PubMed Abstract | CrossRef Full Text | Google Scholar
Estelle, M. (2011). Auxin Signaling: From Synthesis to Systems Biology; a Subject Collection from Cold Spring Harbor Perspectives in Biology. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press.
Google Scholar
, The PB1 domain in auxin response factor and Aux/IAA proteins: a versatile protein interaction module in the auxin response. Plant Cell 27, 33–43. doi: 10.1105/tpc.114.132753
PubMed Abstract | CrossRef Full Text | Google Scholar